1. 领域背景与文献引入
文献英文标题:An ontology for cell types;发表期刊:Genome Biology;影响因子:未公开;研究领域:生物信息学-细胞类型本体论研究
2000年基因本体论(GO)的建立标志着生命科学领域标准化术语体系的重要突破,为基因产物属性注释提供了统一框架,推动了生物数据库的结构化发展。但在模型生物研究领域,表型数据注释、基因表达谱标注等工作仍面临核心瓶颈:缺乏跨物种统一的细胞类型结构化词汇。现有数据库多采用物种特异性的解剖学术语,不同数据库间的细胞类型定义存在差异,无法实现跨平台的数据整合与互操作;同时,多数细胞类型相关资源仅聚焦于细胞的组织归属,缺乏对细胞功能、谱系、表型属性的标准化描述,进一步限制了数据的深度挖掘与复用。针对这一领域空白,本研究旨在构建覆盖原核生物、真菌、动物、植物的跨物种细胞类型本体论,通过标准化的术语定义与多维度分类结构,解决不同物种细胞类型注释的统一性问题,为跨数据库的生物数据整合提供核心支撑。
2. 文献综述解析
作者从覆盖范围、内容维度、可用性三个核心维度,对现有细胞类型相关资源进行系统分类与评述,明确了现有研究的价值与局限性,为跨物种细胞类型本体论的构建奠定了逻辑基础。
现有研究的关键结论包括,基因本体论的成功验证了标准化本体论在生命科学数据整合中的核心价值,物种特异性解剖本体(如小鼠、果蝇解剖本体)能够支持特定模型生物的表型注释,但受限于物种范围无法实现跨平台数据整合;细胞系保藏资源(如ATCC、ECACC)侧重细胞系的存储与分发,缺乏对细胞功能、谱系等表型属性的标准化定义;植物本体仅覆盖植物界细胞类型,无法满足多界生物研究的需求。现有资源的局限性主要体现在三个方面:一是物种覆盖受限,多数资源仅针对单一或少数模型生物,不支持跨物种细胞类型的统一注释;二是信息维度单一,仅聚焦细胞的结构组成或组织归属,未整合功能、谱系等关键属性;三是部分资源未为每个术语分配公开唯一标识符,无法支持自动化的数据库注释与跨平台互操作。对比现有研究的空白,本研究的核心创新在于首次构建覆盖原核、真菌、动物、植物四界的跨物种细胞类型本体论,基于细胞功能、组织学、谱系、倍性等多维度属性定义术语,为每个细胞类型分配唯一公开标识符,并采用有向无环图(DAG)结构支持多维度分类,突破了现有资源的物种与信息维度限制,实现了跨物种细胞类型的标准化与互操作。
3. 研究思路总结与详细解析
本研究以跨物种细胞类型的标准化注释与数据整合需求为导向,整体遵循“需求分析→架构设计→术语构建→验证优化”的闭环逻辑,核心解决跨物种细胞类型同源性争议下的统一分类问题,最终构建了包含680余种细胞类型的多维度本体论体系。
3.1 本体论架构设计与跨物种整合策略确定
本环节的核心目标是确定本体论的整体架构与跨物种细胞类型的整合规则,解决不同物种细胞类型同源性判定的争议问题。
研究团队首先对比了“单物种独立本体”与“跨物种整合本体”两种方案的优劣,明确跨物种整合方案更能满足跨数据库数据互操作的核心需求;针对跨物种细胞类型的同源性争议,团队采用“功能-谱系-进化”三维判定逻辑,对于进化同源性明确的细胞类型(如哺乳动物与线虫的肌细胞)直接归为同一父类,对于同源性存疑的细胞类型(如植物、哺乳动物、昆虫的毛细胞)则添加物种限定词区分。最终确定采用有向无环图(DAG)结构构建本体论,每个术语可通过“is_a”(分类归属)与“develops_from”(谱系来源)两种关系关联多个父类与子类,支持多维度分类检索。
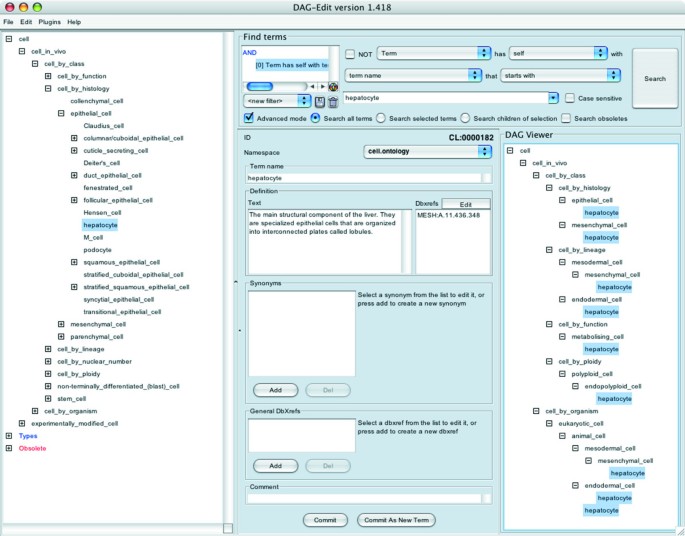
该图片展示了肝细胞在细胞本体论中的多维度分类路径,左侧面板显示肝细胞在组织学分类中的位置,右侧面板展示其所属的所有层级关系,中央面板包含术语定义、唯一标识符等核心信息,直观体现了有向无环图结构的多维度分类优势。文献未提及具体实验产品,领域常规使用本体论构建工具如OBO-Edit、COBrA。
3.2 细胞类型术语库的标准化构建
本环节的核心目标是收集、标准化不同物种的细胞类型术语,构建包含多维度属性的结构化术语库。
研究团队从经典生命科学教材(如《Gray"s Anatomy》《分子细胞生物学》)、权威模型生物数据库(如FlyBase、TAIR)、领域专家咨询等多渠道收集细胞类型信息,最终覆盖原核、真菌、动物、植物四界的680余种细胞类型。对于每个细胞类型,团队按照功能(如电兴奋性细胞、分泌细胞)、组织学(如上皮细胞、间充质细胞)、谱系(如外胚层细胞、内胚层细胞)、倍性(如单倍体细胞、多倍体细胞)等维度进行分类定义,为每个术语分配以“CL:”为前缀的唯一公开标识符,同时添加同义词、文本定义,并关联医学主题词表(MESH)等外部数据库标识符,确保术语的唯一性与可追溯性。结果显示,构建的本体论平均层级深度约为10个节点,每个细胞类型可通过至少2个以上的维度路径进行检索,例如库普弗细胞可同时通过“防御细胞(功能)”“循环细胞(形态)”“动物细胞(物种)”等路径定位。文献未提及具体实验产品,领域常规使用生物数据库资源与本体论编辑工具OBO-Edit。
3.3 本体论实用性与互操作性验证
本环节的核心目标是验证细胞类型本体论在跨数据库数据整合与查询中的实用性。
研究团队设计了三类典型应用场景进行测试:一是跨物种同源基因表达细胞类型查询,如检索小鼠中Notch基因表达的细胞类型,以及果蝇、秀丽隐杆线虫中Notch同源基因表达的细胞类型;二是特定细胞类型的跨物种基因列表查询,如检索小鼠、大鼠、人类、斑马鱼中施万细胞(CL:0000218)表达的基因;三是基于细胞谱系的表型关联查询,如检索果蝇、秀丽隐杆线虫中成肌细胞(CL:0000056)衍生细胞类型中存在突变表型的基因。测试结果表明,该本体论能够支持上述跨物种、多维度的精准查询,实现不同模型生物数据库的数据整合,验证了其在表型注释、基因表达数据标注中的实用性。文献未提及具体实验产品,领域常规使用数据库查询工具与本体论可视化工具COBrA。
4. Biomarker研究及发现成果
本研究中的Biomarker为标准化的细胞类型术语(含唯一标识符),作为生物数据注释与整合的核心“分子标记”,其筛选与验证遵循“多源收集→标准化定义→跨物种验证→唯一标识赋予”的完整逻辑链条。
该类Biomarker的来源覆盖原核、真菌、动物、植物四界的已报道细胞类型,验证方法包括进化同源性分析、多维度属性交叉验证、专家评审确认,确保术语定义的准确性与跨物种适用性。由于本研究属于生物信息学本体论构建范畴,未提供传统Biomarker研究中的特异性、敏感性量化数据(如ROC曲线、AUC值),但每个细胞类型术语均具有唯一公开标识符,支持跨数据库的精准匹配与检索。核心成果方面,该标准化细胞类型术语作为生物数据整合的核心Biomarker,可实现跨物种、跨数据库的基因表达、表型数据的统一注释与查询,其创新性在于首次突破物种限制,基于细胞属性而非组织归属定义Biomarker,采用有向无环图结构支持多维度关联,为生命科学数据的跨平台整合提供了核心支撑。本研究未提供统计学显著性(P值)、样本量等量化数据,所有术语的定义均基于已发表文献与专家共识。
