1. 领域背景与文献引入
文献英文标题:The elusive yeast interactome;发表期刊:Genome Biology;影响因子:未公开;研究领域:酵母蛋白质组与蛋白质相互作用(系统生物学)
蛋白质复合物是细胞功能执行的核心单元,其系统解析是系统生物学的核心研究方向之一。领域关键节点包括1989年酵母双杂交技术的发明(首次实现二元蛋白质相互作用的高通量筛选),2002年大规模串联亲和纯化(TAP)技术的应用(开启了模式生物蛋白质复合物的规模化鉴定)。当前研究热点聚焦于全面解析模式生物的完整互作组,明确蛋白质复合物的功能模块与动态调控机制,但未解决的核心问题包括:不同实验方法得到的互作组数据集重叠度极低,缺乏对实验流程与数据处理方法对结果影响的系统分析,且现有研究仅覆盖了理想生长状态下的细胞,未涉及环境胁迫下的互作组动态变化。
针对上述领域空白,本文以2006年两项最全面的酵母蛋白质复合物大规模研究为对象,系统对比了其数据集差异,解析了方法学层面的原因,明确了当前互作组研究的技术瓶颈,为后续标准化的系统生物学研究提供了关键参考依据。
2. 文献综述解析
本文的综述评述逻辑以“实验方法演进+研究规模升级”为核心分类维度,将酵母蛋白质复合物研究划分为早期小规模探索阶段与2006年大规模全面解析阶段,同时对比了不同技术路线(串联亲和纯化、酵母双杂交)的研究差异。
现有研究的关键结论包括:2002年的两项小规模研究(Gavin等、Ho等)首次证明了大规模纯化蛋白质复合物的可行性,但由于实验流程差异显著,两个数据集的蛋白质重叠度仅为9%,结果可比性极低;2006年的两项大规模研究(Gavin等、Krogan等)均采用串联亲和纯化技术,覆盖了约60%的酵母蛋白质组,鉴定出数百个蛋白质复合物。技术方法优势方面,串联亲和纯化技术能在接近天然状态下捕获蛋白质复合物,结合质谱鉴定可实现大规模、高灵敏度的蛋白质组分析;局限性则体现在早期研究样本量小、方法标准化程度低,2006年的研究虽方法类似,但数据处理策略不同,且仅覆盖了稳定生长状态下的酵母细胞,未考虑环境因素的影响。
本文的创新价值在于首次系统对比了当前最全面的两个酵母蛋白质复合物数据集,揭示了实验流程细节(如跨膜蛋白标签策略、污染蛋白去除规则)与数据处理算法(社会亲和指数SAI vs 机器学习聚类)对结果的显著影响,明确了当前互作组研究中“方法学差异导致结果分歧”的核心问题,为后续研究的方法标准化提供了重要依据,填补了领域内缺乏大规模数据集系统对比的空白。
3. 研究思路总结与详细解析
本文的整体研究框架为:以两项大规模酵母蛋白质复合物研究的数据集为基础,通过对比分析明确结果差异,解析方法学层面的原因,进一步与二元相互作用数据(酵母双杂交)对比揭示不同技术的互补性,最终提出未来互作组研究的方向。核心科学问题是“为何采用类似技术的大规模研究得到的蛋白质复合物数据集差异显著”,技术路线遵循“数据收集→对比分析→方法学解析→跨技术验证→未来展望”的逻辑闭环。
3.1 数据集收集与基础特征分析
本环节的核心目标是获取可对比的大规模蛋白质复合物数据集,明确两项研究的基础覆盖特征。方法细节为收集Gavin等(2006)的原始串联亲和纯化数据与Krogan等(2006)的计算处理后复合物数据,统计蛋白质覆盖度、复合物数量等核心指标。结果解读显示,Gavin等的研究鉴定出491个蛋白质复合物,覆盖1483个蛋白质(占酵母蛋白质组的23%);Krogan等的研究鉴定出547个蛋白质复合物,覆盖2702个蛋白质(占酵母蛋白质组的42%);两个数据集的重叠蛋白质仅1152个(占酵母蛋白质组的18%),基础特征差异显著。文献未提及具体实验产品,领域常规使用串联亲和纯化标签试剂盒、高分辨率质谱仪等。
3.2 复合物重叠度与方法学差异解析
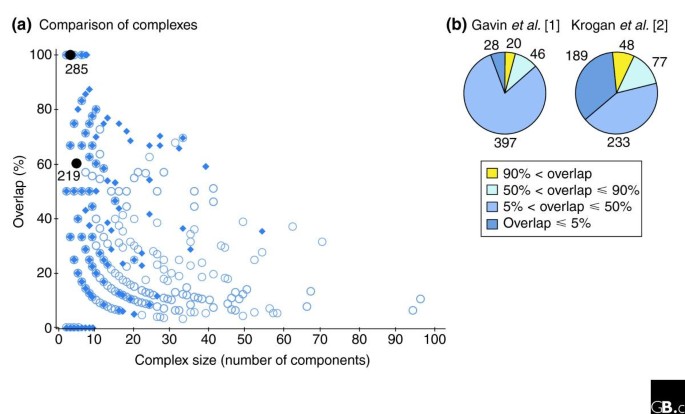
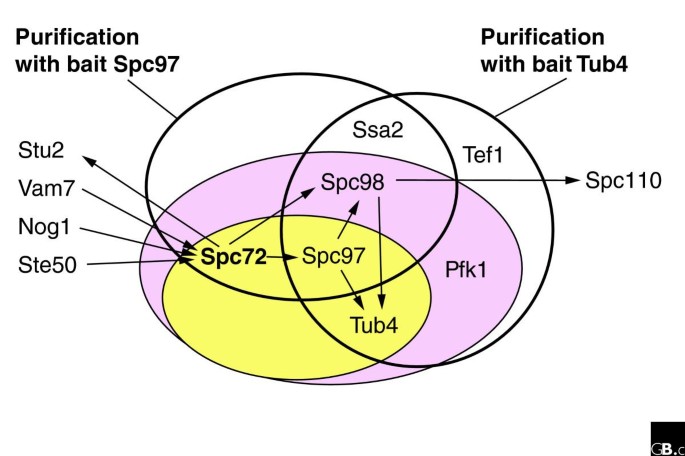
本环节的核心目标是解析两项研究数据集差异的关键原因。方法细节为对比两项研究的实验流程差异(Gavin等尝试标记所有跨膜蛋白,Krogan等去除了出现频率超过3%的非特异性污染蛋白)与数据处理算法差异(Gavin等基于原始数据发明社会亲和指数SAI定义复合物,Krogan等采用机器学习算法聚类得到复合物)。结果解读显示,两项研究中仅6个复合物完全相同,Gavin等定义的27.62%核心复合物被包含在Krogan等定义的21.02%复合物中,差异主要源于实验流程的细节差异与数据处理的算法选择,其中数据处理对复合物的最终定义影响最为显著。
3.3 与二元相互作用数据的交叉对比
本环节的核心目标是揭示不同蛋白质相互作用研究技术的互补性。方法细节为将两项串联亲和纯化研究的复合物数据与已发表的酵母双杂交数据集进行对比,分析两种技术捕获的相互作用类型差异。结果解读显示,串联亲和纯化技术主要捕获稳定存在的多亚基蛋白质复合物,而酵母双杂交技术更多捕获瞬时的二元蛋白质相互作用,两者的数据集重叠度极低,仅少数小分子量复合物存在部分重叠,证明了不同技术在蛋白质相互作用研究中的互补性。
3.4 研究局限与未来方向梳理
本环节的核心目标是明确当前酵母互作组研究的未解决问题与未来方向。方法细节为基于现有研究的局限性,分析环境因素对互作组的影响、数据整合的需求等。结果解读显示,现有研究仅覆盖了理想生长状态下的酵母细胞,而自然环境中酵母常处于胁迫状态,蛋白质复合物的组成与相互作用会发生显著变化;同时,领域内缺乏统一的数据处理标准与整合框架,未来需要开展环境依赖的动态互作组研究,建立标准化的数据分析流程,实现多组学数据的整合解析。
4. Biomarker研究及发现成果
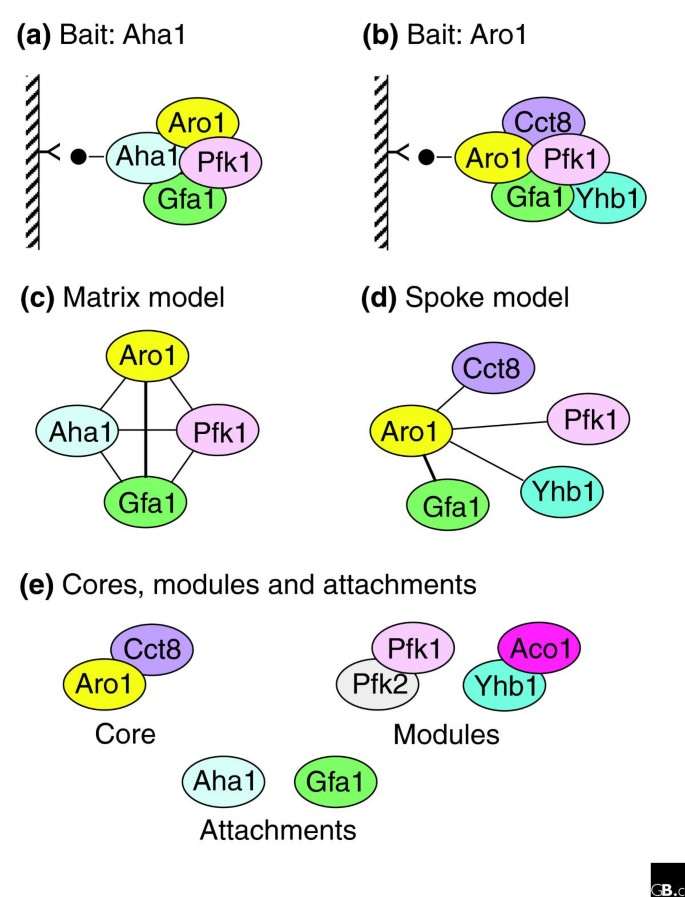
本文中涉及的Biomarker类型为“酵母功能蛋白质复合物模块”,其筛选与验证逻辑为:基于大规模串联亲和纯化数据,通过社会亲和指数(SAI)或机器学习算法对共纯化蛋白质进行聚类,得到具有稳定功能的核心复合物、可调节的功能模块与松散结合的附件蛋白,形成“核心-模块-附件”的三级结构体系。
研究过程详述:Biomarker的来源为酵母细胞内的天然蛋白质复合物,验证方法为串联亲和纯化结合质谱鉴定,通过重复实验验证复合物的稳定性(Gavin等重复了139次纯化实验,69%的蛋白质在重复实验中被共纯化);特异性方面,Gavin等的研究覆盖了73%的已知酵母蛋白质复合物,新发现257个未报道的复合物,SAI值高的蛋白质对具有更高的共纯化稳定性(文献未提供具体敏感性与特异性的量化数据)。
核心成果提炼:首次定义了酵母蛋白质复合物的“核心-模块-附件”功能结构,揭示了不同实验方法与数据处理策略对复合物鉴定的显著影响,为系统生物学中功能模块的研究提供了范式;创新性在于首次系统对比了两项最全面的酵母复合物数据集,明确了技术方法对结果的调控作用;统计学结果显示,Gavin等的重复实验中69%的蛋白质重复出现(n=139,文献未提供P值),两项研究的蛋白质重叠度为18%(文献未提供P值)。该成果为后续模式生物互作组的标准化研究奠定了基础,推动了系统生物学领域对功能模块的认知。
