1. 领域背景与文献引入
文献英文标题:Functional Impact of RNA editing and ADARs on regulation of gene expression: perspectives from deep sequencing studies;发表期刊:Cell & Bioscience;影响因子:未公开;研究领域:RNA编辑与基因表达调控
领域共识:RNA编辑是转录后基因表达调控的核心环节之一,其中腺苷-肌苷(A-to-I)编辑是后生动物中最主要的类型,由作用于RNA的腺苷脱氨酶(ADAR)家族催化。早期研究(20世纪90年代)发现ADAR介导的A-to-I编辑可改变RNA编码信息,如谷氨酸受体基因的编辑影响受体功能;2004年左右,基于公共序列数据的生物信息学分析首次揭示人类转录组中广泛存在Alu元件的A-to-I编辑;下一代测序(NGS)技术的兴起(2010年后)推动了RNA编辑组的全局解析,实现了单核苷酸分辨率的编辑位点鉴定。当前研究热点聚焦于RNA编辑与其他转录后调控机制的交互作用,包括可变剪接、转录本稳定性、微小RNA(miRNA)功能等,但核心问题仍未解决:RNA编辑如何与这些机制协同调控基因表达,以及是否存在统一的“RNA编辑代码”指导其功能发挥。
针对这一研究空白,本文系统整合了近年基于NGS技术的RNA编辑研究成果,全面解析RNA编辑与可变剪接、转录本稳定性及miRNA生物学的交互调控网络,为建立RNA编辑代码、深入理解基因表达调控的复杂性提供了重要框架。
2. 文献综述解析
作者以RNA编辑与不同转录后调控机制的交互为分类维度,系统综述了NGS技术应用以来RNA编辑领域的研究进展,重点梳理了现有研究的局部证据与全局分析的差异,明确了当前研究的局限性与未来方向。
早期研究主要聚焦于个别基因的RNA编辑与可变剪接的关联,如ADAR2通过编辑自身前体mRNA产生功能缺失的剪接异构体,形成负反馈调控;谷氨酸受体B(Gria2)基因的R/G编辑位点可影响剪接效率,这些研究证实了编辑对剪接的直接调控,但仅覆盖少量基因,缺乏全局视角。转录本稳定性与定位方面,早期假设A-to-I编辑可通过p54^{nrb}依赖机制使异常转录本滞留于细胞核,但后续研究存在矛盾,部分实验显示编辑与核滞留无显著关联,缺乏全局转录本的验证数据。miRNA生物学领域,早期通过靶向RT-PCR发现个别miRNA的编辑可改变其靶向特异性,但未揭示miRNA编辑组的全局特征及与RNA编辑的双向调控关系。现有研究的技术方法优势在于NGS技术可实现单核苷酸分辨率的全局编辑组分析,但局限性在于部分研究存在测序误差、比对偏差等技术问题,且多数研究仅关注单一调控机制,缺乏多组学的整合分析。
与现有研究相比,本文的创新点在于首次系统整合了不同NGS研究的数据,全面解析了RNA编辑与可变剪接、转录本稳定性、miRNA的双向调控网络,提出了建立“RNA编辑代码”的可能性,填补了对RNA编辑与其他转录后调控机制协同作用的认知空白,为后续全局研究提供了统一的分析框架。
3. 研究思路总结与详细解析
本文为综述性研究,整体研究目标是系统梳理基于NGS技术的RNA编辑与基因表达调控交互机制的研究进展,核心科学问题是RNA编辑如何通过与可变剪接、转录本稳定性、miRNA的协同作用调控基因表达,技术路线为分类整合已发表的NGS相关研究数据,解析各调控模块的交互证据,最后提出未来研究方向与挑战。
3.1 RNA编辑与可变剪接调控的关联分析
实验目的:解析RNA编辑对可变剪接的调控机制,明确顺式与反式调控的作用模式。
方法细节:梳理已发表的高通量RNA-seq、外显子芯片分析等研究,包括ADAR1敲低后的转录组分析、剪接调控元件的编辑位点鉴定等。
结果解读:全局研究显示ADAR1敲低后,数百个基因的剪接模式发生改变;部分基因的剪接调控元件(如外显子内的剪接调控序列)存在直接的A-to-I编辑,少数情况下编辑可改变经典剪接位点(受体、供体、分支位点),但这类直接调控并非ADAR1影响剪接的主要机制;更多的是ADAR1通过调控剪接相关反式作用因子的表达,间接影响可变剪接,且受影响的基因富集于RNA剪接与加工功能。
产品关联:文献未提及具体实验产品,领域常规使用高通量测序平台(如Illumina NovaSeq系列)、RNA干扰试剂、剪接分析生物信息学工具(如rMATS)等。
3.2 RNA编辑对转录本稳定性与定位的调控解析
实验目的:明确RNA编辑对转录本稳定性与细胞定位的调控作用,解决早期研究的矛盾结论。
方法细节:整合核与胞质转录组测序、蛋白质免疫共沉淀等研究数据,分析编辑位点在转录本中的分布特征,以及ADAR与RNA结合蛋白的相互作用。
结果解读:NGS研究显示编辑位点显著富集于转录本的3"非翻译区(3"UTR),提示编辑可能调控转录本稳定性;ADAR1与RNA结合蛋白HuR存在相互作用,且编辑位点附近富集HuR结合序列,证实两者协同调控转录本加工;核与胞质编辑组的对比研究显示,编辑状态与转录本核滞留无显著关联,否定了早期的核滞留假设。
产品关联:文献未提及具体实验产品,领域常规使用核质分离试剂盒、蛋白质免疫共沉淀试剂盒、转录组测序分析工具等。
3.3 RNA编辑与miRNA生物学的双向调控研究
实验目的:解析RNA编辑与miRNA的双向调控关系,明确其对基因表达的影响。
方法细节:梳理小RNA-seq、转录组测序等研究数据,分析miRNA前体与成熟miRNA的编辑位点,以及3"UTR编辑位点与miRNA结合位点的重叠情况。
结果解读:全局miRNA编辑组分析显示,miRNA前体与成熟序列中存在广泛的A-to-I编辑,部分编辑位于种子区,可改变miRNA的靶向特异性,如miR-376的编辑可重定向其靶向基因;同时,3"UTR的A-to-I编辑可破坏或创建miRNA结合位点,如ARHGAP26基因3"UTR的编辑可干扰miR-30b-3p与miR-573的结合,从而调控基因表达;此外,研究还发现miRNA序列中存在非ADAR介导的A-to-G编辑,提示存在其他编辑机制。
产品关联:文献未提及具体实验产品,领域常规使用小RNA测序平台、miRNA靶标预测工具(如TargetScan)、qRT-PCR验证试剂等。
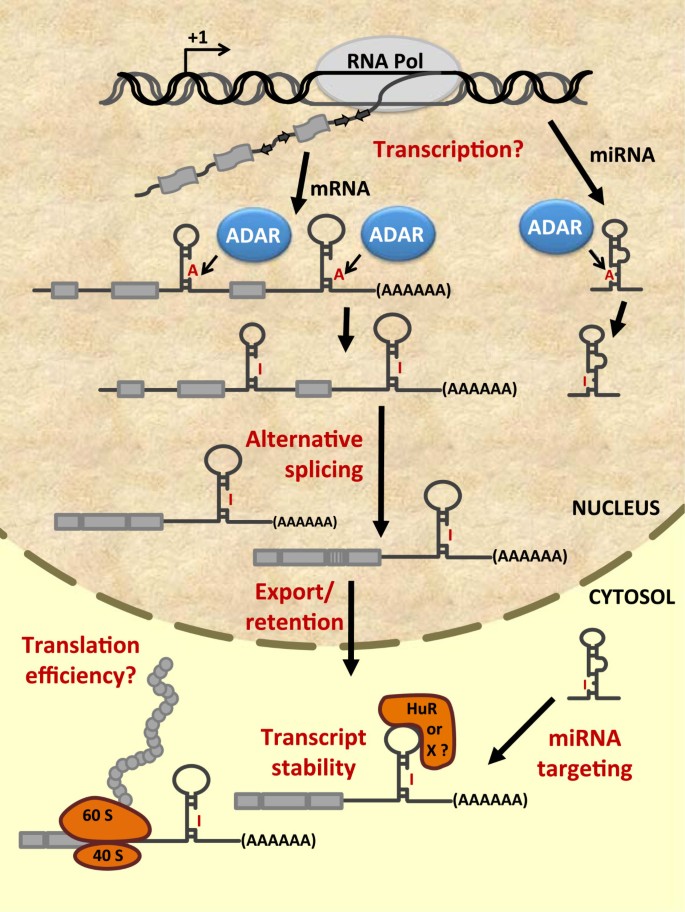
该图系统展示了ADAR与RNA编辑对细胞RNA种类的调控模式,涵盖可变剪接、转录本稳定性、miRNA生物发生与功能等多个环节,直观呈现了RNA编辑与多种转录后调控机制的交互网络,为理解基因表达调控的复杂性提供了可视化框架。
3.4 RNA编辑在转录调控中的潜在作用探讨
实验目的:探讨RNA编辑在转录调控中的潜在功能,补充对其全层面调控作用的认知。
方法细节:梳理ADAR结构域分析、基因敲低后的转录组分析、代谢标记测序等研究数据,分析ADAR与转录调控因子的相互作用及对转录活性的影响。
结果解读:ADAR1含有Z-DNA结合域,提示其可能与染色质区域结合参与转录调控;ADAR1可与转录调控因子NF90相互作用,调控其基因表达功能;GLI1基因转录本的A-to-I编辑可改变其编码的氨基酸序列,从而影响其转录活性与细胞生长功能;但现有研究仅能提供稳态转录本水平的变化,缺乏直接的转录合成速率数据,需代谢标记测序(如4sU-Seq)、全局核运行测序(GRO-Seq)等技术进一步验证。
产品关联:文献未提及具体实验产品,领域常规使用染色质免疫共沉淀试剂盒、代谢标记试剂、转录组测序分析工具等。
4. Biomarker研究及发现成果
本文为综述性研究,未涉及特定生物标志物(Biomarker)的筛选、验证及功能研究,主要聚焦于RNA编辑与基因表达调控机制的交互分析,为后续疾病相关Biomarker的研究提供了理论基础。
综述中提及ADAR1突变或A-to-I编辑异常与多种人类疾病相关,包括肿瘤恶性进展、遗传性对称性色素异常症、散发性肌萎缩侧索硬化(ALS)、神经精神疾病等,提示疾病相关的RNA编辑位点可作为潜在的诊断或预后Biomarker。推测:基于本文提出的RNA编辑与其他调控机制的交互网络,结合临床样本的NGS数据,有望筛选出具有高特异性与敏感性的RNA编辑Biomarker,其筛选逻辑可遵循“数据库筛选→细胞系验证→临床样本验证”的链条,验证方法可包括定量PCR、数字PCR、RNA编辑组测序等,为疾病的早期诊断与预后评估提供新的靶点。目前尚无针对RNA编辑Biomarker的大规模临床验证数据,未来研究需进一步明确其特异性、敏感性及统计学特征(如ROC曲线AUC值、风险比HR等),以推动其临床转化应用。
