1. 领域背景与文献引入
文献英文标题:Tunable PhenoCycler imaging of the murine pre-clinical tumour microenvironments;发表期刊:Cell & Bioscience;影响因子:未公开;研究领域:肿瘤微环境空间生物学、临床前小鼠模型高复用成像
过去20年,肿瘤微环境(TME)在肿瘤进展、免疫逃逸及治疗响应中的核心作用逐渐被认知,领域核心认知从仅关注肿瘤细胞的致癌突变,发展到重视TME中免疫细胞、内皮细胞、成纤维细胞等非肿瘤成分与肿瘤细胞的互作。当前研究热点聚焦于TME的空间结构对免疫功能的调控,以及空间表型与治疗响应的关联,但现有技术存在明显局限:多参数流式细胞术是TME免疫表型分析的主流方法,可实现高通量细胞分群,但需解离组织,完全丢失细胞的空间位置信息;传统免疫荧光(IF)成像能保留组织架构,但通常仅能检测1-2种标记,无法全面解析复杂TME的细胞组成。近10年高复用组织成像技术快速兴起,其中PhenoCycler(原CODEX)平台可实现高达101种标记的单细胞空间表型分析,已在人多种肿瘤及非肿瘤组织中广泛应用,但针对临床前小鼠模型的应用局限于新鲜冰冻组织,而大量科研团队存档的临床前研究组织以福尔马林固定石蜡包埋(FFPE)形式保存,这一方法学空白限制了存档资源的再利用,也增加了重复造模的成本与伦理负担。本文旨在开发适用于小鼠FFPE组织的可调PhenoCycler抗体面板及开源数据分析流程,利用存档组织开展TME空间表型分析,为临床前肿瘤研究提供高效的空间生物学工具。
2. 文献综述解析
作者对领域内现有研究的分类维度包括技术类型(流式细胞术、传统免疫荧光、高复用成像)、组织保存形式(新鲜冰冻vs FFPE)、研究物种(人vs小鼠),通过多维度对比梳理现有研究的优势与局限,凸显本研究的方法学创新价值。
现有研究的关键结论可分为三类,一是流式细胞术作为TME免疫表型分析的金标准,能精准量化免疫细胞亚群的比例,但无法提供细胞间的空间互作信息,难以解析TME的结构功能关联;二是传统免疫荧光成像可保留组织的空间架构,但标记数有限,无法同时解析TME中多种细胞类型的互作;三是PhenoCycler等高复用成像技术已在人皮肤T细胞淋巴瘤、结直肠癌、黑色素瘤等多种肿瘤及溃疡性结肠炎、阿尔茨海默病等非肿瘤组织中应用,实现了单细胞水平的TME空间表型解析,但针对小鼠组织的应用仅局限于新鲜冰冻样本,FFPE小鼠组织的高复用成像缺乏标准化的抗体优化、偶联及成像流程。现有技术的优势在于,流式细胞术的高通量细胞分群能力、PhenoCycler的高标记数空间成像能力;局限性则体现在流式的空间信息丢失、传统IF的低标记数、小鼠FFPE组织高复用成像的方法学空白。本研究的创新价值在于,首次建立并验证了适用于小鼠FFPE组织的可调PhenoCycler抗体面板,构建了从抗体筛选、寡核苷酸barcode偶联到成像及开源数据分析的完整流程,填补了小鼠FFPE组织高复用空间成像的方法学空白,为利用存档临床前组织开展空间生物学研究提供了可行方案。
3. 研究思路总结与详细解析
本研究的整体框架以“方法学建立-样本验证-生物学发现”为闭环,研究目标是开发小鼠FFPE组织的PhenoCycler高复用成像方案并应用于多种肿瘤模型的TME空间分析,核心科学问题包括如何实现小鼠FFPE组织的高特异性高复用成像、不同肿瘤模型TME的空间组成及细胞互作差异,技术路线涵盖抗体筛选与验证、抗体偶联与优化、FFPE组织染色成像、开源数据分析、多模型TME比较分析五个关键环节。
3.1 抗体筛选与免疫荧光验证
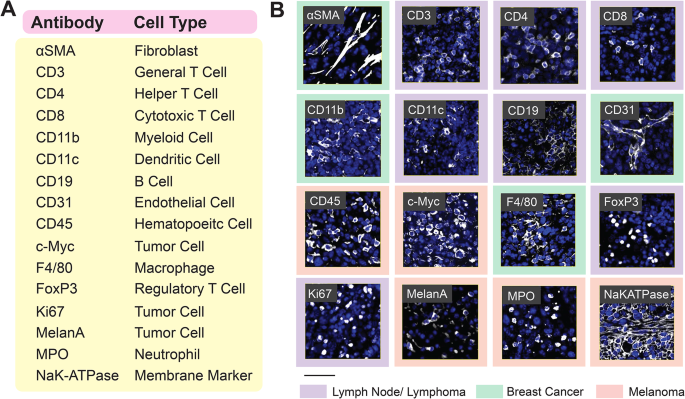
该环节的核心目标是筛选适用于小鼠FFPE组织的抗体克隆,验证其染色特异性与信噪比(SNR)。方法细节为取4μm厚的小鼠FFPE组织切片,依次进行脱蜡(二甲苯或HistoChoice替代)、梯度乙醇复水、抗原修复(Tris-EDTA缓冲液pH9.0,90℃处理20min),随后进行双重封闭:先用Primary Blocking Buffer室温封闭30min,再用FC Blocking Buffer室温封闭30min;一抗以10μg/mL浓度(约1:100稀释)在4℃湿度箱中孵育过夜,次日加入AlexaFluor647标记的二抗室温孵育1h,最后用DAPI复染细胞核,使用与PhenoCycler配套的Fusion显微镜成像。结果解读显示,通过与ProteinAtlas数据库的染色模式对比,结合病理学家的评估,最终筛选出16种抗体,可覆盖TME中的主要细胞类型,包括肿瘤细胞(Ki67、c-Myc)、内皮细胞(CD31)、成纤维细胞(αSMA)、髓系细胞(CD11b、F4/80、CD11c、MPO)、淋系细胞(CD3、CD4、CD8、FoxP3、CD19),所有抗体的染色模式符合预期,SNR满足后续寡核苷酸barcode偶联的要求。产品关联:文献未提及具体实验产品,领域常规使用抗小鼠免疫细胞、基质细胞及肿瘤细胞标记的单克隆抗体,以及免疫荧光染色相关缓冲液与封闭试剂。
3.2 抗体与寡核苷酸Barcode偶联及验证
该环节的核心目标是将筛选后的抗体与PhenoCycler特异性寡核苷酸barcode偶联,并验证偶联效率。方法细节为使用Akoya Biosciences的抗体偶联试剂盒,先将抗体通过50kDa MWCO柱脱盐去除载体蛋白,随后加入抗体还原混合液室温处理30min,再加入预制备的barcode溶液室温孵育2h,通过三次纯化去除未结合的barcode,最后将偶联抗体洗脱至抗体储存液中;通过10% SDS-PAGE凝胶电泳验证偶联效率,对比未偶联抗体与偶联抗体的分子量迁移差异。结果解读显示,偶联抗体的分子量较未偶联抗体明显上移,表明barcode偶联成功;根据前期免疫荧光实验的SNR结果,为不同抗体匹配了对应的荧光报告基团:SNR高的抗体偶联AF750标记的报告基团,低丰度抗原的抗体偶联AF647标记的报告基团,中等丰度抗原的抗体偶联ATTO550标记的报告基团,以确保成像信号的均衡性。产品关联:实验所用关键产品:Akoya Biosciences的PhenoCycler抗体偶联试剂盒、寡核苷酸barcode及荧光报告基团试剂。
3.3 FFPE组织PhenoCycler染色与成像优化
该环节的核心目标是优化偶联抗体的染色条件,确保PhenoCycler成像的特异性与信噪比。方法细节为FFPE切片脱蜡、抗原修复后,用含H₂O₂的漂白溶液进行两次各45min的LED光漂白,以淬灭FFPE组织的自发荧光;随后用PhenoCycler染色缓冲液平衡组织30min,加入稀释后的偶联抗体,优化孵育条件(3h室温或4℃过夜);手动加入对应荧光报告基团孵育5min,洗涤后成像,对比传统免疫荧光的染色结果评估成像质量。结果解读显示,优化后的染色条件下,PhenoCycler成像的信号强度与染色模式与传统免疫荧光完全一致,SNR良好,无明显非特异性染色;最终建立的16重抗体面板可同时标记TME中的所有主要细胞类型,满足后续空间分析的需求。产品关联:实验所用关键产品:Akoya Biosciences的PhenoCycler染色缓冲液、封闭试剂、荧光报告基团。
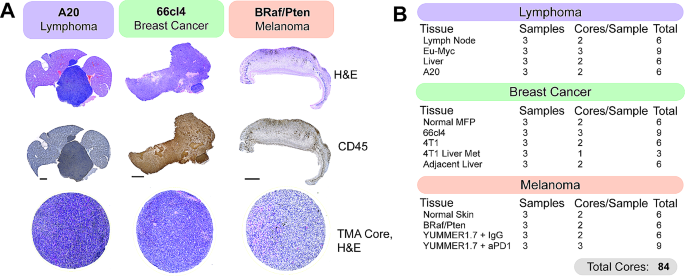
3.4 多癌种小鼠组织芯片(TMA)构建
该环节的核心目标是构建包含多种肿瘤模型的TMA,用于批量PhenoCycler成像分析,减少实验误差并提高效率。方法细节为选取淋巴瘤(A20结外淋巴瘤、Eµ-Myc nodal淋巴瘤)、乳腺癌(66cl4低转移乳腺癌、4T1高转移原发乳腺癌及肝转移灶)、黑色素瘤(BRAFV600E/PTEN-/-免疫冷黑色素瘤、YUMMER1.7免疫热黑色素瘤的IgG对照及αPD-1处理组)的FFPE组织块,先进行H&E和CD45染色,筛选免疫细胞富集的肿瘤区域,使用组织芯片制作仪取1mm直径的组织芯,构建包含84个芯的多癌种TMA。结果解读显示,该TMA覆盖了不同肿瘤类型、不同转移状态、不同免疫治疗响应的样本,为后续TME的空间表型比较分析提供了标准化的实验材料,避免了单个样本成像的系统误差。产品关联:文献未提及具体实验产品,领域常规使用组织芯片制作仪、H&E染色试剂盒、抗CD45单克隆抗体。
3.5 PhenoCycler多复用成像与开源数据分析
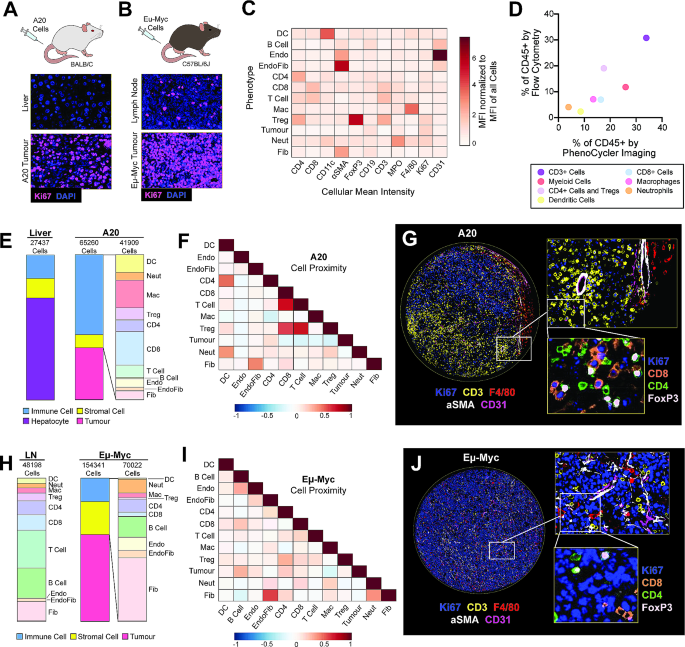
该环节的核心目标是对TMA样本进行PhenoCycler多复用成像,并通过开源流程实现细胞分割、分类及空间互作分析。方法细节为使用PhenoCycler-Fusion系统进行自动化成像,每个循环检测3种不同荧光标记的抗体,完成所有16种标记的成像后,系统自动进行图像拼接与背景校正,生成QPTIFF格式的多复用图像;使用开源软件QuPath v3.2进行图像分析,通过StarDist插件实现基于DAPI的单细胞分割,手动标注30-60个目标细胞及对照细胞,训练人工神经网络分类器,将细胞分为10种表型;使用CytoMAP软件进行空间邻域分析,计算50μm半径内不同细胞类型的Pearson相关系数,评估细胞间的空间互作。结果解读显示,PhenoCycler成像成功实现了所有TMA样本的16种标记同时检测,细胞分类结果与存档的流式细胞术数据高度相关(A20淋巴瘤的相关性r=0.8551,P=0.0142,文献未明确样本量,基于图表趋势推测),验证了细胞分类的准确性;空间邻域分析揭示了不同细胞类型的空间聚集或排斥关系,为解析TME的结构功能关联提供了量化依据。产品关联:实验所用关键产品:Akoya Biosciences的PhenoCycler-Fusion成像系统;开源分析软件QuPath v3.2、CytoMAP。
3.6 不同肿瘤模型TME的空间表型分析
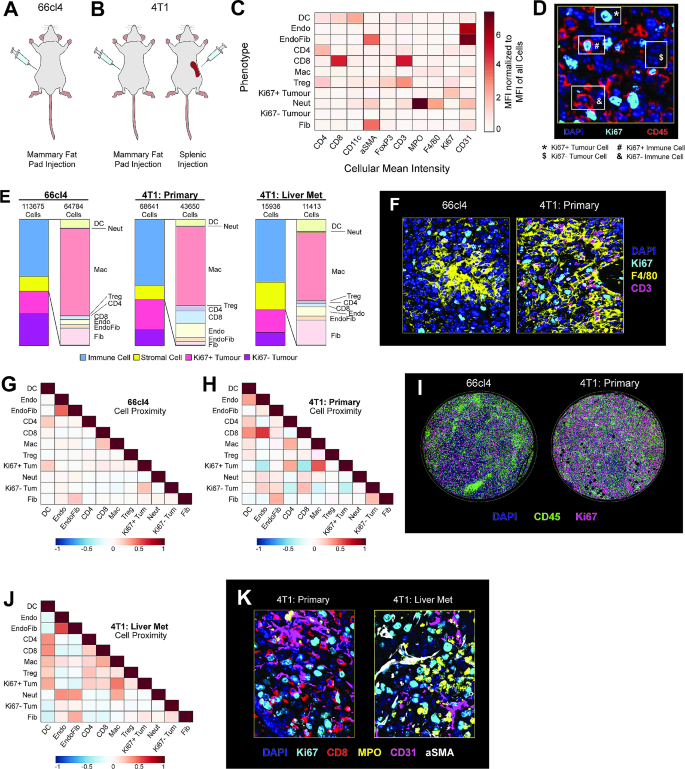
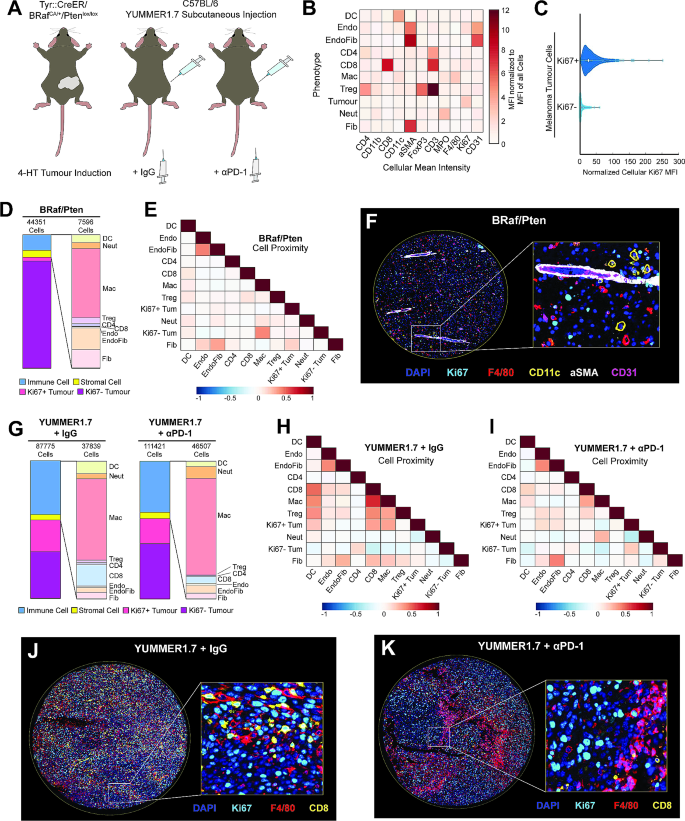
该环节的核心目标是比较不同肿瘤模型、不同转移状态、不同免疫治疗响应样本的TME细胞组成与空间互作差异。方法细节为通过QuPath统计每个样本中不同细胞表型的比例,通过CytoMAP计算细胞间的空间相关系数,生成热图展示空间互作关系,并结合成像结果可视化TME的结构特征。结果解读显示,淋巴瘤模型中,A20结外淋巴瘤的免疫细胞浸润率高达55.12%(文献未明确样本量,基于图表趋势推测),远高于相邻正常肝组织的16.27%(文献未明确样本量,基于图表趋势推测),且调节性T细胞(Treg)与CD8+T细胞的空间相关系数为0.695(文献未明确样本量,基于图表趋势推测),提示二者紧密互作,而肿瘤细胞与CD8+T细胞的相关系数为-0.1289(文献未明确样本量,基于图表趋势推测),提示肿瘤细胞与免疫细胞空间分离;Eµ-Myc nodal淋巴瘤的免疫细胞浸润率仅为16.41%(文献未明确样本量,基于图表趋势推测),远低于正常淋巴结的84.04%(文献未明确样本量,基于图表趋势推测),细胞间无明显空间互作关系。乳腺癌模型中,高转移4T1原发肿瘤的Ki67+增殖肿瘤细胞比例为80.65%(文献未明确样本量,基于图表趋势推测),显著高于低转移66cl4肿瘤的41.13%(文献未明确样本量,基于图表趋势推测);4T1肝转移灶中中性粒细胞与内皮细胞的空间相关系数为0.4064(文献未明确样本量,基于图表趋势推测),显著高于原发肿瘤的0.19%(文献未明确样本量,基于图表趋势推测),提示中性粒细胞与内皮细胞的互作可能促进肝转移。黑色素瘤模型中,BRAFV600E/PTEN-/-免疫冷肿瘤的免疫细胞浸润率仅为11.83%(文献未明确样本量,基于图表趋势推测),细胞分布随机;YUMMER1.7免疫热肿瘤经αPD-1治疗耐药后,CD8+T细胞与Ki67+肿瘤细胞的空间相关系数从0.3529(文献未明确样本量,基于图表趋势推测)降至无显著相关性,提示肿瘤细胞与免疫细胞的空间互作丢失是耐药的重要机制。产品关联:文献未提及具体实验产品,领域常规使用上述开源分析软件进行空间生物学数据解析。
4. Biomarker研究及发现成果
本文涉及的Biomarker为肿瘤微环境中的细胞表型组成及空间互作特征,筛选逻辑是基于多癌种小鼠TMA的PhenoCycler成像数据,通过细胞分类与空间分析,鉴定与肿瘤类型、转移状态、免疫治疗响应相关的量化指标,为临床前研究的治疗靶点筛选提供空间维度的依据。
Biomarker的来源为小鼠FFPE肿瘤组织TMA样本,验证方法为PhenoCycler高复用成像结合QuPath细胞分类与CytoMAP空间分析,特异性通过传统免疫荧光验证,敏感性通过与流式细胞术数据的相关性验证(r=0.8551,P=0.0142,文献未明确样本量,基于图表趋势推测)。核心成果包括三类Biomarker:一是肿瘤类型相关Biomarker,结外B淋巴瘤(A20)中Treg与CD8+T细胞的空间相关系数(0.695,文献未明确样本量,基于图表趋势推测)可作为免疫抑制的空间Biomarker,而nodal B淋巴瘤(Eµ-Myc)中免疫细胞浸润率降低(16.41%,文献未明确样本量,基于图表趋势推测)可作为免疫耗竭的Biomarker;二是转移相关Biomarker,4T1乳腺癌肝转移灶中中性粒细胞与内皮细胞的空间相关系数(0.4064,文献未明确样本量,基于图表趋势推测)及中性粒细胞比例(0.88%,文献未明确样本量,基于图表趋势推测)可作为肝转移的Biomarker;三是免疫治疗耐药相关Biomarker,YUMMER1.7黑色素瘤αPD-1耐药后,CD8+T细胞与Ki67+肿瘤细胞的空间互作丢失可作为免疫治疗耐药的Biomarker。创新性在于首次在小鼠FFPE组织中通过高复用成像鉴定了TME的空间Biomarker,填补了临床前研究中空间Biomarker的空白,为肿瘤治疗的临床前评估提供了新的量化指标。统计学结果显示,所有空间相关系数均标注Pearson相关系数及P值,细胞比例数据基于多个TMA芯的统计(文献未明确单个模型的样本量,基于图表趋势推测)。
