1. 领域背景与文献引入
文献英文标题:Identification of a novel Rev-interacting cellular protein;发表期刊:BMC Cell Biology;影响因子:未公开;研究领域:HIV-1 Rev蛋白与细胞因子相互作用机制。
人类免疫缺陷病毒(HIV)复制的核心步骤依赖于Rev蛋白——作为转录后激活因子,Rev通过结合病毒mRNA的Rev响应元件(RRE),促进未剪接/部分剪接mRNA的核输出,是HIV产生子代病毒的关键调控因子。Rev的结构具有模块化特征:N端精氨酸富集基序(ARM)兼具核定位信号(NLS)和RNA结合功能;中间区域介导多聚化;C端亮氨酸富集区是核输出信号(NES)。截至本研究开展时,已发现多种细胞因子与Rev相互作用(如ARM结合的p32、B23;C端结合的CRM1、核孔蛋白eIF-5A等),但Rev与细胞因子的相互作用网络仍未完全阐明,新型相互作用因子的功能及调控机制仍是研究空白。
本研究针对“Rev相互作用细胞因子的全面性不足”这一问题,通过酵母双杂交筛选鉴定新型Rev相互作用蛋白,旨在解析其对Rev功能的调控机制,为HIV抗病毒治疗提供潜在靶点。
2. 文献综述解析
文献综述围绕“Rev的核心功能”与“已发现的相互作用因子”展开核心评述:作者首先强调Rev在HIV复制中的不可替代性——其核质穿梭和mRNA调控功能直接决定病毒颗粒的生成;随后按结合结构域分类总结已发现的相互作用因子:(1)结合ARM域的因子(如p32、B23),分别参与抑制病毒mRNA剪接、促进Rev核输入;(2)结合C端NES域的因子(如CRM1、核孔蛋白),介导Rev的核输出;(3)其他因子(如REBP),参与Rev的活性调控。现有研究的局限性在于:对Rev与细胞因子相互作用的全面性不足,尤其是新型因子的功能未知,且缺乏对因子间协同作用的系统解析。
本研究的创新价值在于:通过酵母双杂交从T细胞文库中筛选到新型Rev相互作用蛋白16.4.1(后命名为Risp),并首次系统阐明其与Rev的相互作用区域、亚细胞定位及对Rev功能的调控机制,补充了Rev的细胞相互作用网络。
3. 研究思路总结与详细解析
本研究以“鉴定新型Rev相互作用细胞蛋白并解析其功能”为目标,采用“筛选-验证-定位-功能”的闭环思路:通过酵母双杂交筛选候选蛋白,再经分子生物学技术验证相互作用、解析亚细胞定位,最终探究其对Rev功能的影响。
3.1 酵母双杂交筛选Rev相互作用蛋白
实验目的:从T细胞cDNA文库中筛选与HIV-1 Rev特异性相互作用的细胞蛋白。
方法细节:以全长Rev为诱饵(pEG202-sRev)转化酵母EGY48(含pSH18-34报告质粒),再转染Jurkat T细胞cDNA文库(pJG4-5载体)。通过“葡萄糖抑制/半乳糖诱导”的选择性培养基筛选,结合β-半乳糖苷酶活性检测鉴定阳性克隆。
结果解读:筛选获得46个阳性克隆,经测序和复筛,得到两个特异性相互作用克隆——11.5.1(DNA结合蛋白dbpB片段)和16.4.1。后续聚焦16.4.1分析,其与Rev的相互作用依赖半乳糖诱导,且不与反义Rev、LexCD2等对照诱饵结合,表明相互作用具有特异性。
产品关联:文献未提及具体实验产品,领域常规使用酵母双杂交系统(如Clontech Matchmaker Gold)、Jurkat T细胞cDNA文库(如Stratagene)及酵母选择性培养基(Yeast Nitrogen Base)。
3.2 相互作用区域定位(Rev与16.4.1的关键区域)
实验目的:确定Rev与16.4.1相互作用的核心氨基酸区域。
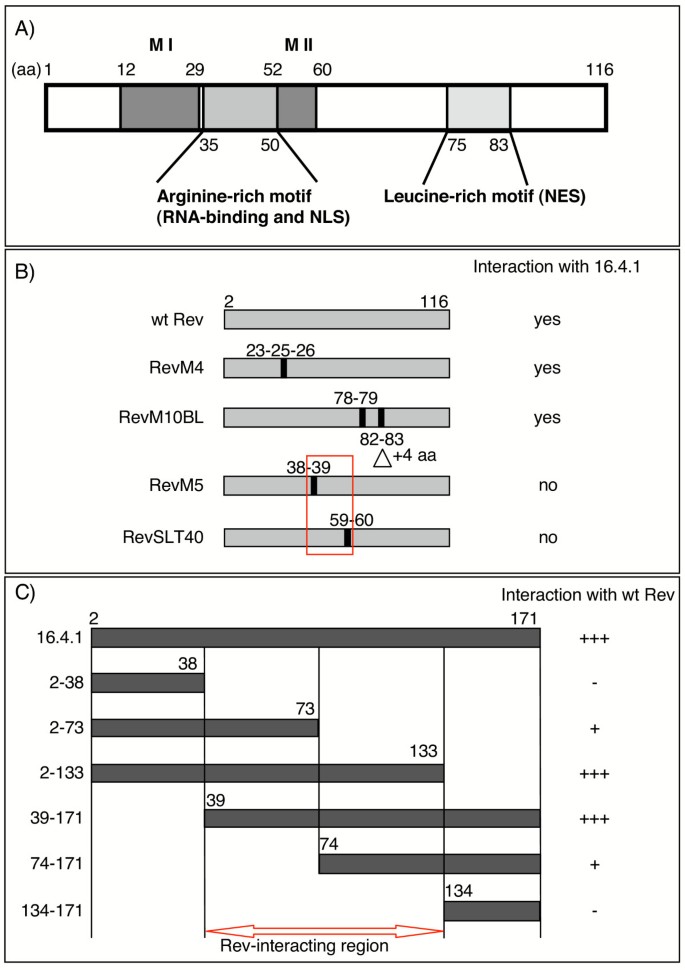
方法细节:(1)Rev突变体分析:构建Rev功能域突变体(RevM4:多聚化域;RevM5:ARM域;RevSLT40:多聚化域;RevM10BL:NES域),作为诱饵与16.4.1 prey杂交,通过酵母生长情况判断相互作用;(2)16.4.1截短体分析:构建16.4.1的N端、C端及中间片段prey,与Rev bait杂交。
结果解读:(1)Rev的38-60aa区域(ARM+多聚化域)是相互作用关键——RevM5(ARM域R38/R39突变)、RevSLT40(多聚化域I59/L60突变)无法与16.4.1结合,而RevM4(多聚化域)、RevM10BL(NES域)仍能结合(图1);(2)16.4.1的39-133aa区域介导与Rev的相互作用——该片段与Rev的结合效率与全长一致,而N端(2-38aa)或C端(134-171aa)片段无相互作用(图1)。
产品关联:文献未提及具体实验产品,领域常规使用PCR克隆试剂盒(如TaKaRa PrimeSTAR)、酵母转化试剂(如Zymogen Frozen-EZ Yeast Transformation II)。
3.3 哺乳动物细胞中的相互作用验证
实验目的:验证16.4.1与Rev、CRM1在人细胞中的相互作用,及16.4.1的同源多聚化。
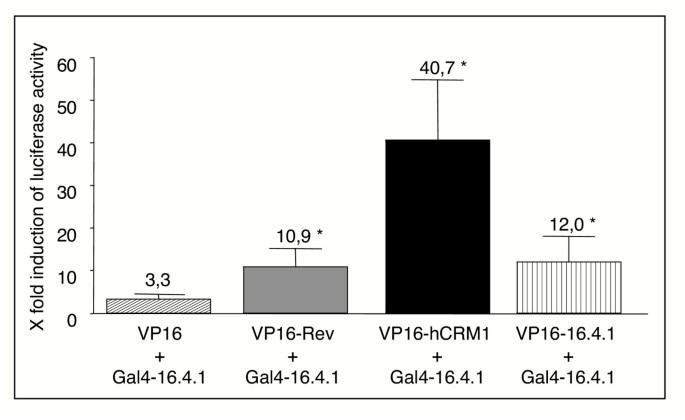
方法细节:采用Promega CheckMate哺乳动物双杂交系统,构建Gal4-16.4.1(pBIND-16.4.1)与VP16-Rev、VP16-CRM1、VP16-16.4.1(pACT载体),共转染HEK293细胞,检测荧光素酶活性。
结果解读:(1)Gal4-16.4.1与VP16-Rev共转染诱导荧光素酶活性约11倍(n=14,p<0.04);(2)与VP16-CRM1共转染诱导约41倍(n=7,p<0.04);(3)与VP16-16.4.1共转染诱导约12倍(n=6,p<0.04),均显著高于对照(3.3倍),表明16.4.1在人细胞中与Rev、CRM1相互作用,并形成同源多聚体(图2)。
产品关联:文献提及使用Promega CheckMate™ Mammalian Two-Hybrid System,其他未明确,领域常规使用Lipofectamine 2000转染试剂、Bright-Glo荧光素酶检测试剂盒。
3.4 16.4.1的亚细胞定位及CRM1依赖性
实验目的:分析16.4.1的亚细胞定位及是否依赖CRM1介导的核输出。
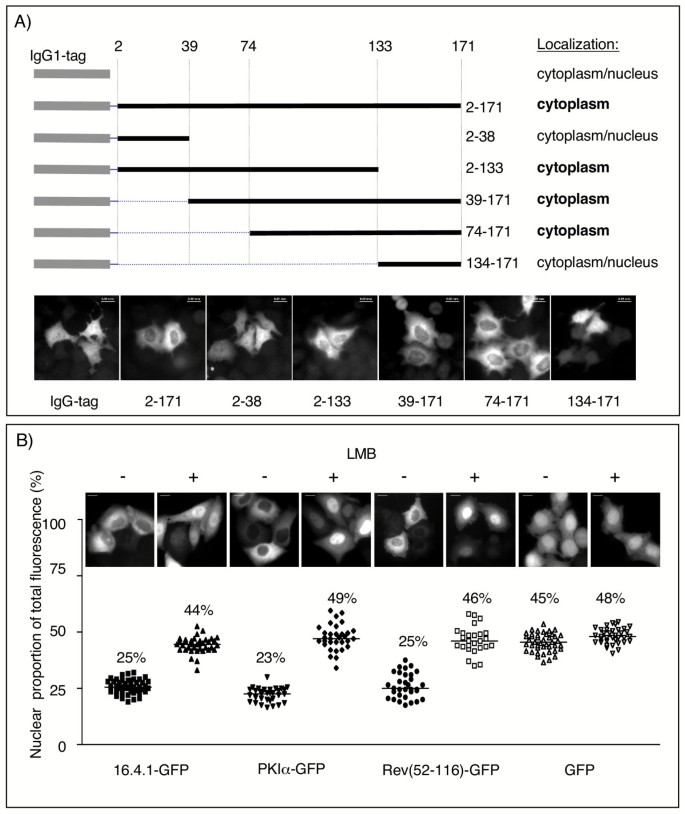
方法细节:(1)构建16.4.1的IgG1/GFP融合蛋白(如pC16.4.1sg143),转染HeLa细胞,通过免疫荧光(IgG1-16.4.1用Cy3标记二抗)或直接荧光观察定位;(2)用CRM1抑制剂Leptomycin B(LMB,5nM处理2h)处理,定量分析核荧光比例。
结果解读:(1)全长16.4.1-GFP及截短体(2-133、39-171、74-171aa)主要定位于细胞质,而N端(2-38aa)或C端(134-171aa)片段核质均有分布,表明74-133aa区域介导胞质定位(图3A);(2)LMB处理后,16.4.1-GFP的核荧光比例从25%升至44%,与已知CRM1底物(PKIα-GFP、Rev(52-116)-GFP)的变化一致,表明16.4.1的胞质定位依赖CRM1(图3B)。
产品关联:文献未提及具体实验产品,领域常规使用Jackson ImmunoResearch Cy3标记二抗、Sigma-Aldrich LMB、Zeiss Axiovert荧光显微镜。
3.5 16.4.1核输出信号(NES)的鉴定
实验目的:定位16.4.1中的NES并验证其功能。
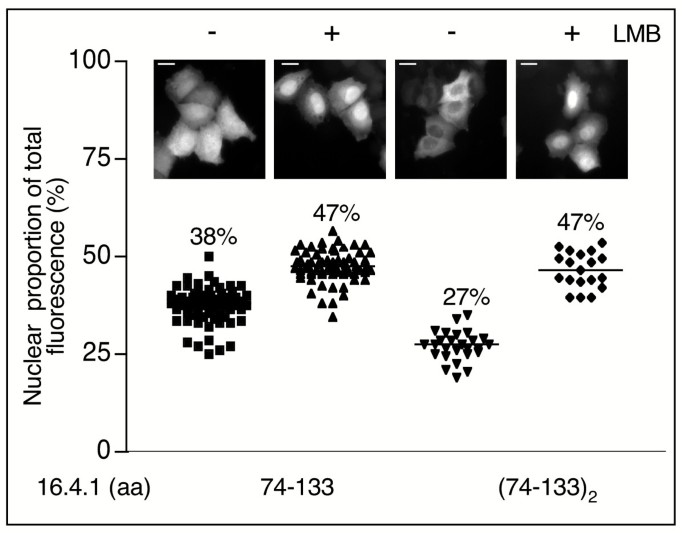
方法细节:(1)构建16.4.1的GFP截短体(74-133aa单拷贝/双拷贝),分析定位;(2)合成16.4.1的86-105aa肽段,与Alexa-red标记BSA偶联,核微注射后检测核输出;(3)生物信息学分析NES序列(用NESbase和MatInspector)。
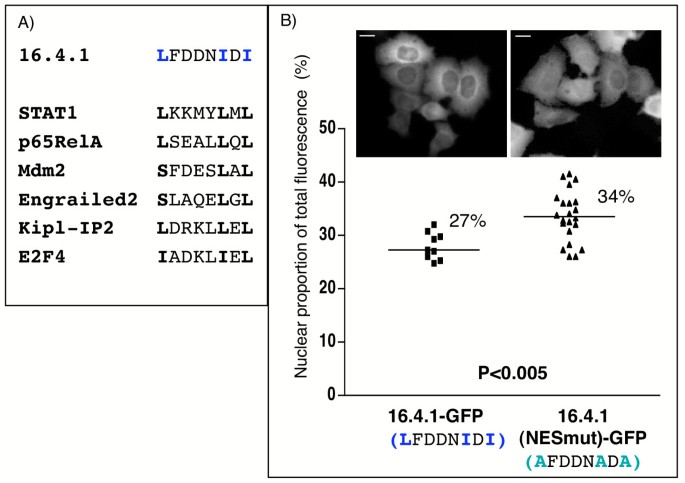
结果解读:(1)74-133aa双拷贝的胞质定位强于单拷贝,表明该区域含NES;(2)86-105aa肽段的核输出活性与Rev NES相当(相对translocation活性<1),表明该区域是功能性NES(图4、5);(3)生物信息学分析显示86-105aa含Leu/Ile富集区,与Stat1、p65RelA的NES类似,但不同于Rev或PKIα的NES(图6A);(4)突变NES的Leu/Ile(L92A、I97A、I99A)后,核荧光比例从27%升至34%,表明NES是胞质定位的关键但非唯一因素(图6B)。
产品关联:文献未提及具体实验产品,领域常规使用Sigma-Genosys肽合成服务、Eppendorf FemtoJet微注射系统、NCBI BLAST/Genomatix MatInspector生物信息学工具。

3.6 Rev与16.4.1的共定位分析
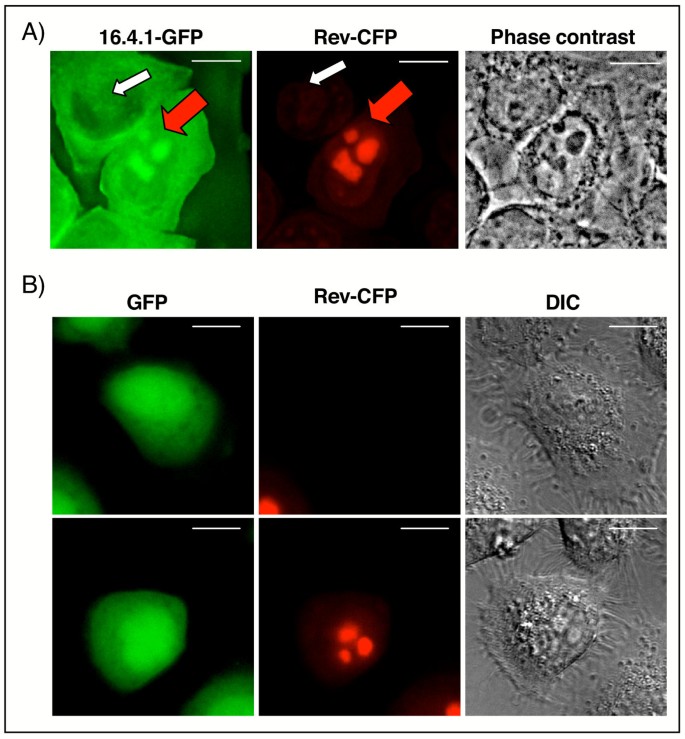
实验目的:验证Rev是否能改变16.4.1的亚细胞定位,探究两者在细胞内的相互作用位点。
方法细节:构建稳定表达16.4.1-GFP的HeLa细胞系(HeLa 16.4.1-GFP),转染Rev-CFP表达质粒,通过Zeiss Axiovert 200M 3D共聚焦显微镜观察,结合去卷积和多通道解混技术分析共定位。
结果解读:仅在共表达Rev-CFP的细胞中,16.4.1-GFP定位于核仁;单独表达16.4.1-GFP时仅在细胞质,Rev-CFP保持核/核仁定位,表明Rev能招募16.4.1至核仁(图7)。
产品关联:文献未提及具体实验产品,领域常规使用Zeiss LSM共聚焦显微镜、G418细胞系筛选试剂。
3.7 16.4.1对Rev转录激活功能的影响
实验目的:探究16.4.1过表达对Rev转录激活功能的调控作用。
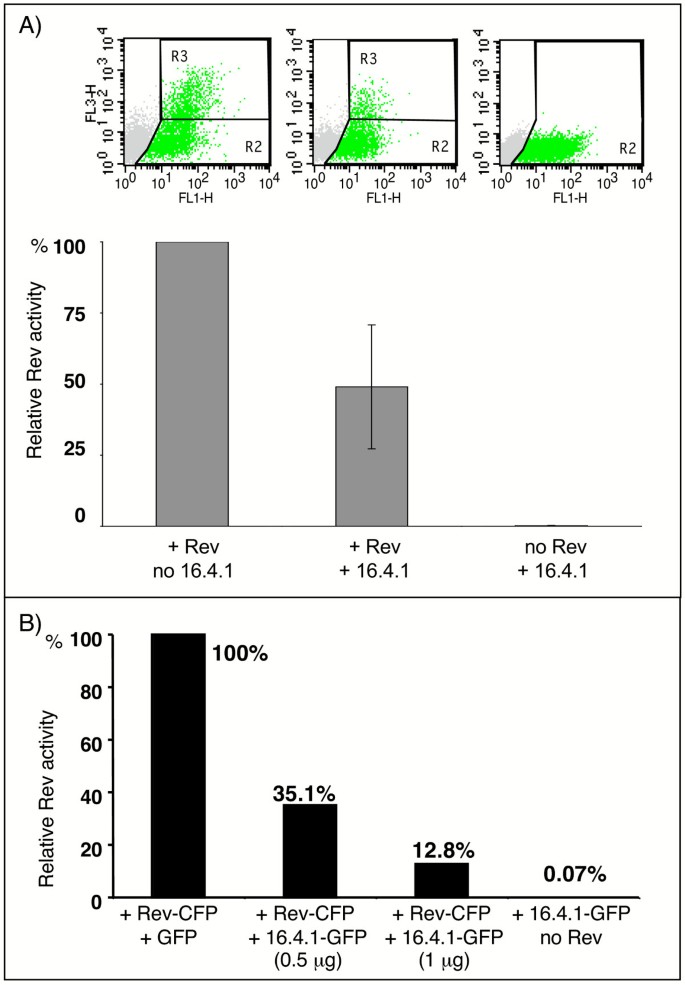
方法细节:采用Rev报告系统(pLRed(INS)₂R,Rev/Tat依赖的RFP表达),转染293T细胞,共转染16.4.1-GFP或Rev-CFP,通过BD FACSCalibur流式细胞术分析RFP阳性细胞比例(Rev活性指标)。
结果解读:16.4.1过表达使Rev活性从100%降至约50%(5次独立实验,图8A),且呈剂量依赖性——0.5μg质粒使活性降至35.1%,1μg降至12.8%(图8B),表明16.4.1抑制Rev的转录激活功能。
产品关联:文献未提及具体实验产品,领域常规使用BD FACSCalibur流式细胞仪、pGL3系列报告质粒。
3.8 RNAi介导的内源性16.4.1功能验证
实验目的:验证内源性16.4.1对Rev功能的调控作用。
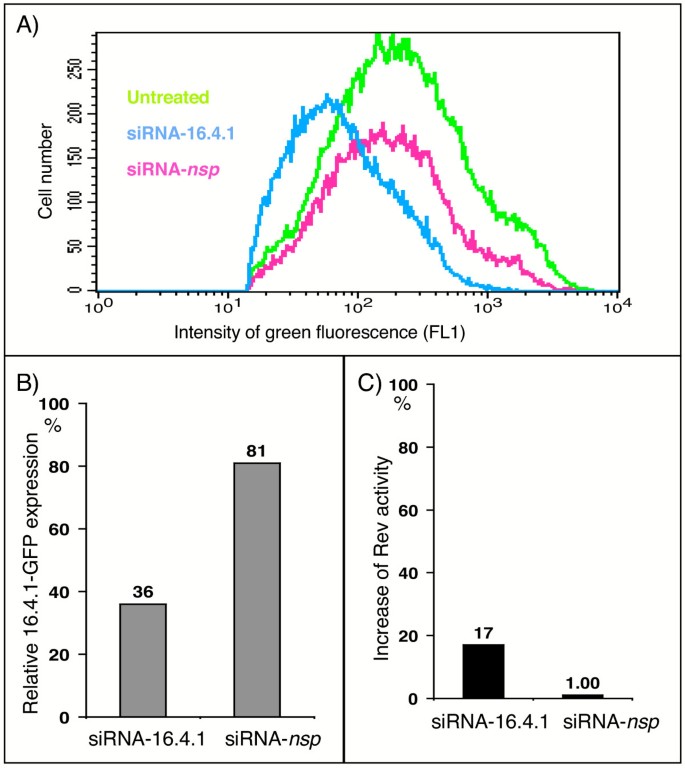
方法细节:设计16.4.1特异性siRNA(siRNA-16.4.1)和非特异性对照(siRNA-nsp),转染293T细胞,24h后转染Rev报告系统,通过FACS分析Rev活性;同时转染HeLa 16.4.1-GFP细胞,验证siRNA的沉默效率(FACS检测GFP表达)。
结果解读:(1)siRNA-16.4.1使16.4.1-GFP表达降至36%(对照siRNA-nsp为81%,图9A、B);(2)siRNA-16.4.1使Rev活性增强17%,而siRNA-nsp无显著影响(图9C),表明内源性16.4.1抑制Rev功能。
产品关联:文献提及使用Qiagen siRNA合成服务,其他未明确,领域常规使用Qiagen RNAiFect转染试剂。
4. Biomarker研究及发现成果解析
本研究鉴定的16.4.1(Risp)是Rev功能调控性Biomarker,属于细胞内蛋白Biomarker,其筛选与验证逻辑为“酵母双杂交筛选→细胞系相互作用验证→功能调控验证”。
Biomarker定位与研究过程
- 来源:Jurkat T细胞cDNA文库;
- 验证方法:酵母双杂交(选择性生长)、哺乳动物双杂交(荧光素酶活性)、共定位(免疫荧光)、功能实验(Rev报告系统);
- 特异性:仅与Rev的38-60aa区域相互作用,不与其他对照诱饵结合;
- 敏感性:siRNA敲低16.4.1后,Rev活性增强17%(n=3,p<0.04);过表达使活性降低50%(n=5,p<0.04)。
核心成果提炼
16.4.1是首次鉴定的新型Rev相互作用细胞蛋白,其功能与调控机制具有以下创新性:
1. 相互作用机制:通过与Rev的ARM+多聚化域(38-60aa)相互作用,被Rev招募至核仁;
2. 亚细胞定位:依赖CRM1介导的核输出定位于细胞质,其74-133aa区域含新型NES(86-105aa);
3. 功能调控:过表达抑制Rev的转录激活功能,敲低则增强,是Rev功能的负调控因子。
该Biomarker的价值在于补充了Rev的细胞相互作用网络——首次揭示了Rev与新型细胞因子的相互作用及调控机制,为理解HIV复制的细胞调控提供了新节点。潜在应用方向为抗病毒治疗靶点:通过抑制16.4.1可增强Rev功能(但需结合HIV复制的整体调控,实际可能作为抑制Rev功能的靶点,阻断病毒mRNA核输出)。
本研究通过系统解析16.4.1与Rev的相互作用及功能,为HIV-1 Rev蛋白的细胞调控研究提供了新线索,也为抗病毒治疗的靶点开发奠定了基础。
