1. 领域背景与文献引入
文献英文标题:High resolution imaging reveals heterogeneity in chromatin states between cells that is not inherited through cell division;发表期刊:BMC Cell Biology;影响因子:未公开;研究领域:染色质结构与表观遗传
染色质作为DNA-蛋白质复合物,是真核生物基因组的存在形式,其构象动态直接调控基因转录等核心生物学过程。领域发展关键节点包括1979年Thoma等发现组蛋白H1参与染色质高级结构组装,2004年Dorigo等提出染色质30nm纤维的双起始模型,2009年Lieberman-Aiden等通过Hi-C技术首次绘制全基因组染色体相互作用图谱。当前研究热点聚焦于染色质构象的动态调控机制、细胞间异质性的产生及功能,以及表观遗传状态的跨代传递。然而,现有研究仍存在核心空白:一方面,Hi-C等捕获技术依赖化学交联,无法实时观察染色质构象的动态变化;另一方面,染色质构象的细胞间异质性是否可通过细胞分裂遗传,以及哪些未知因素调控染色质压缩状态,尚未得到直接的实验证据。针对这一空白,本研究通过建立高分辨率活细胞成像系统,直接解析了染色质构象的动态变化、细胞间异质性及遗传特性,为理解细胞间转录异质性的起源提供了新视角。
2. 文献综述解析
作者对领域内现有研究的分类维度主要包括技术手段(生化重构、生物物理分析、染色体构象捕获技术、活细胞成像)和研究结论(染色质结构模型、构象动态特性、表观遗传继承)。
现有研究的关键结论显示,生化重构实验已明确核小体的基本结构,生物物理研究提出了30nm染色质纤维的单起始螺线管、双起始超螺旋等多种模型,但体内研究仅在少数特殊细胞类型中观察到规则的30nm纤维,多数情况下染色质以无规则核小体阵列形式存在;染色体构象捕获技术(如Hi-C)揭示了染色体拓扑结构域(TAD)的存在,但这类技术依赖DNA交联,无法捕获染色质构象的时间动态信息;活细胞成像技术可实时追踪染色质位点的运动,但早期研究的测量精度有限,难以精准解析构象异质性。技术方法方面,Hi-C等技术的优势在于能提供全基因组范围的染色体相互作用图谱,活细胞成像的优势在于能实时观察染色质的动态变化;局限性则表现为Hi-C无法反映构象的时间动态,传统活细胞成像的测量误差较大,且针对染色质构象异质性遗传特性的研究较为匮乏。
与现有研究相比,本研究的创新价值在于首次建立了测量精度达63nm的活细胞3D染色质成像 workflow,突破了传统成像的精度限制;直接观察到染色质构象的细胞间异质性可维持数分钟,但不通过细胞分裂遗传;同时发现已知的染色质特征(如组蛋白修饰、转录因子结合、染色体相互作用)与染色质压缩状态无显著相关性,提示存在未知调控因素,填补了染色质构象动态与遗传研究的空白。
3. 研究思路总结与详细解析
本研究的整体框架为:以酿酒酵母为模式生物,构建荧光标记的染色体质点追踪模型,建立高分辨率活细胞成像系统,测量不同基因组区间的染色质压缩状态,分析染色质构象的时间动态变化与细胞间异质性,最终验证染色质构象异质性的细胞分裂遗传特性。核心科学问题聚焦于染色质构象异质性的调控机制及跨细胞分裂的遗传能力,技术路线遵循“模型构建→精度优化→特性分析→遗传验证”的闭环逻辑。
3.1 荧光标记酵母菌株构建与高分辨率成像系统建立
实验目的是构建可实时追踪染色质构象的活细胞模型,并建立精度足够的成像分析流程,以准确测量染色质位点的3D位置。方法细节上,研究人员构建了7株酿酒酵母菌株,在不同长度的基因组区间两侧分别整合256个lac操纵子阵列和224个tet操纵子阵列,通过GFP标记的LacI蛋白和mCherry标记的TetR蛋白特异性结合操纵子,实现对目标染色质位点的荧光标记;采用GE|OMX Blaze®显微镜进行活细胞3D成像,成像流程包含暗场减噪、ND-SAFIR算法去噪、Softworx软件去卷积等步骤,并通过两步通道对齐策略——首先利用多波长荧光珠子进行粗对齐,再通过表达共定位荧光标记的对照菌株进行细对齐——将位点距离测量误差从110nm降至63nm。结果解读显示,成功建立的成像系统可实现活细胞中染色质位点的3D追踪,63nm的测量精度足以捕捉染色质构象的细微变化。产品关联:文献未提及具体实验产品,领域常规使用荧光蛋白标记的转录抑制因子、高分辨率活细胞结构光照明显微镜及专业图像去噪与分析软件。
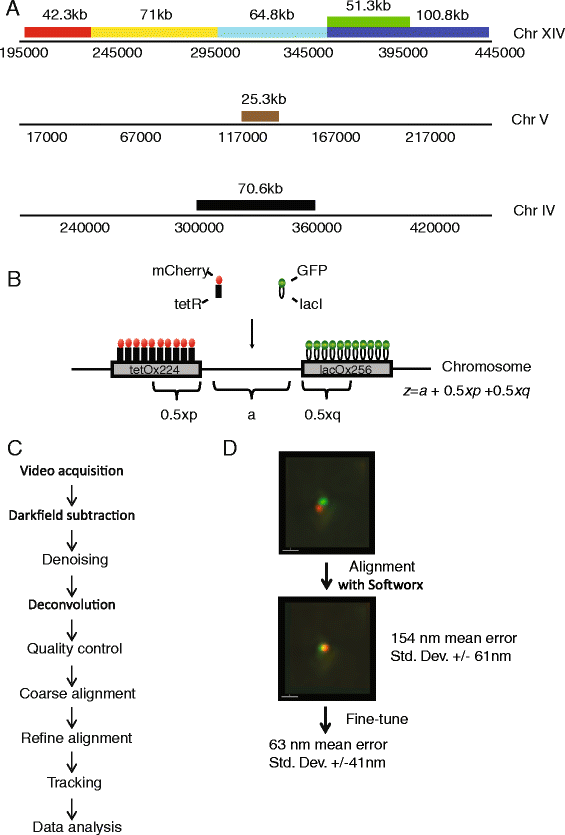
3.2 染色质压缩状态与基因组区间的关联分析
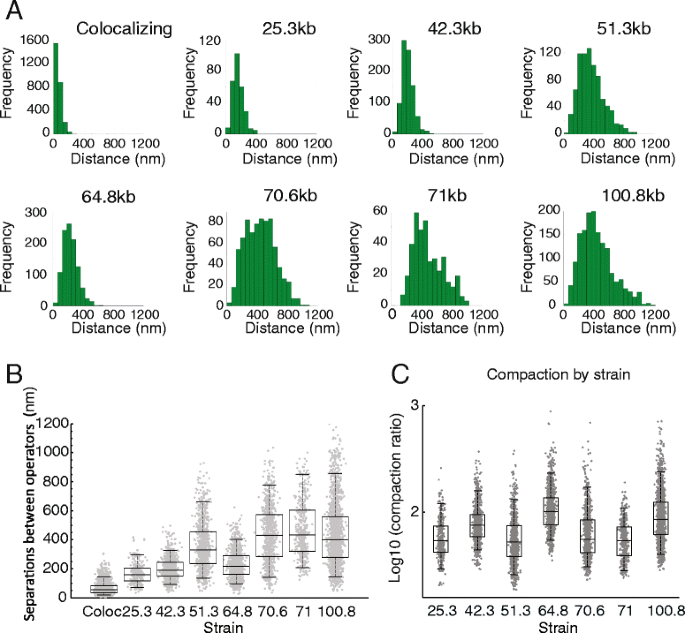
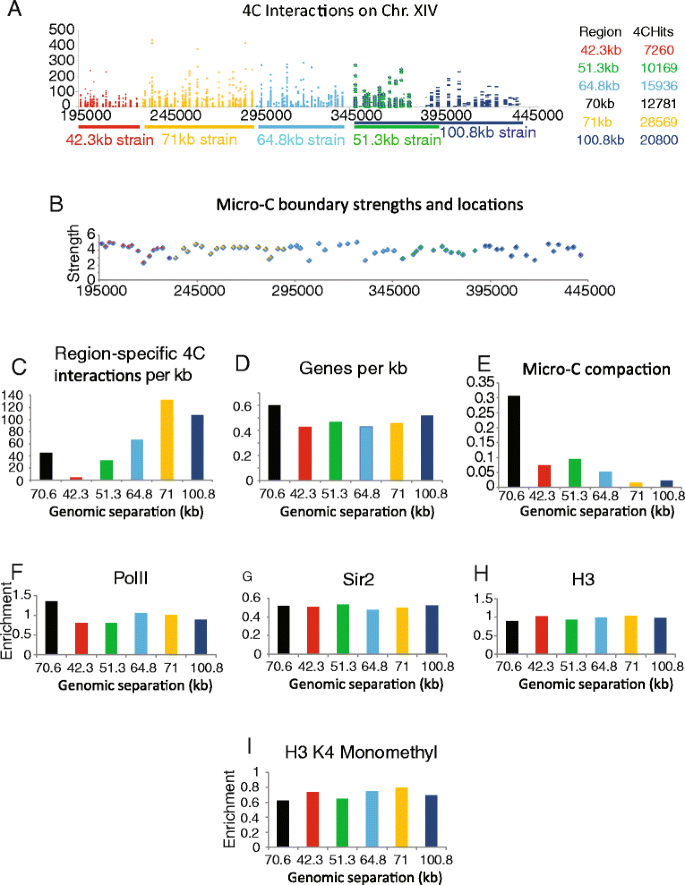
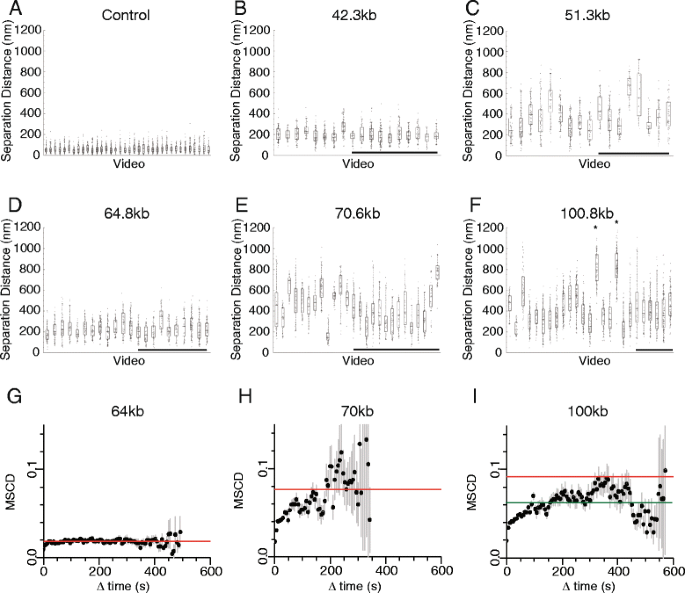
实验目的是解析不同基因组区间的染色质压缩特性,并探索调控染色质压缩的潜在因素。方法细节上,研究人员对7株菌株在细胞周期G1期的荧光位点物理距离进行测量,计算染色质压缩比(基因组长度与物理距离的比值);同时结合已发表的Hi-C、Micro-C染色体相互作用数据,以及18种染色质特征(组蛋白修饰、转录因子结合、组蛋白 occupancy等)的ChIP-seq数据,分析这些因素与染色质压缩状态的相关性。结果解读显示,染色质位点的物理距离随基因组长度增加呈非线性增长,其中42kb、64kb、100kb基因组区间的染色质压缩比显著高于其他区间;但Hi-C/Micro-C检测的染色体相互作用频率、组蛋白修饰水平、转录因子结合丰度等已知染色质特征,与染色质压缩状态无显著相关性,提示存在尚未鉴定的调控因素影响染色质构象。
3.3 染色质构象的动态变化与细胞间异质性分析
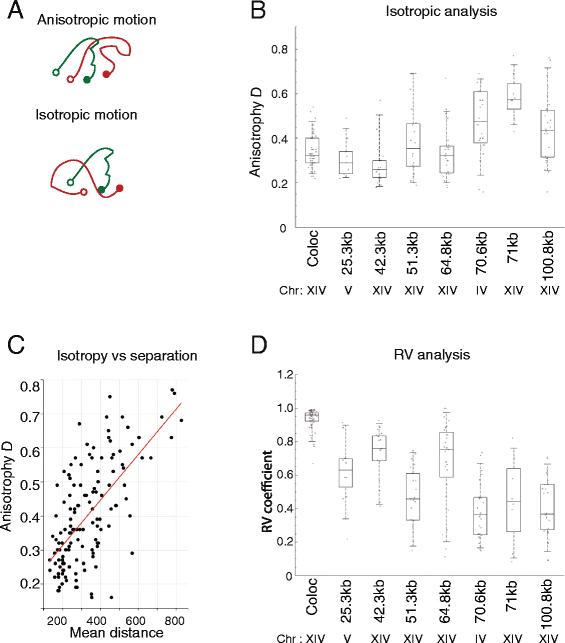
实验目的是研究单个细胞内染色质构象的时间动态变化,以及细胞间染色质构象的异质性特征。方法细节上,研究人员对每个菌株采集了多段包含50-100个时间点的活细胞成像视频,计算荧光位点距离的均方变化(MSCD)以分析构象变化的时间尺度;通过各向异性统计量D评估位点运动的约束程度,利用RV系数分析两个标记位点的运动相关性。结果解读显示,单个细胞内的染色质位点距离随时间持续波动,细胞间的平均位点距离差异可达2-4倍,且这种异质性可在细胞中维持1-10分钟;MSCD曲线在30-150秒后进入平台期,表明染色质构象的变化在该时间尺度后趋于稳定;当基因组分离长度超过70kb时,位点运动的各向异性显著增加,运动相关性降低,提示细胞核环境对远距离染色质位点的运动约束更强。
3.4 染色质构象异质性的遗传特性验证
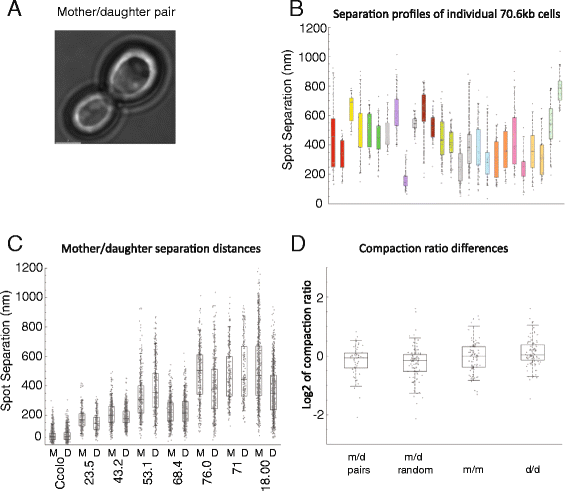
实验目的是明确染色质构象的细胞间异质性是否可通过细胞分裂传递给子代细胞。方法细节上,研究人员利用酿酒酵母母细胞与子细胞在分裂后短暂连接的特性,观察母-子细胞对的染色质位点距离,计算压缩比变化,并将相关母-子对的压缩比变化与随机选取的非相关母-子对进行统计比较(Mann-Whitney检验)。结果解读显示,母细胞与子细胞的染色质位点距离无显著相关性,相关母-子对与非相关母-子对的压缩比变化分布相似(Mann-Whitney p=0.181),表明染色质构象的细胞间异质性不会通过细胞分裂遗传。
4. Biomarker研究及发现成果解析
本研究中涉及的Biomarker类型为染色质构象异质性状态,以荧光标记染色质位点的物理距离为量化指标,其筛选与验证逻辑为:通过高分辨率活细胞成像筛选不同细胞的染色质构象差异→时间序列分析验证异质性的维持时间→母-子细胞对比验证遗传特性,形成完整的研究链条。
该Biomarker的来源为酿酒酵母活细胞内的染色质位点,验证方法为高分辨率3D活细胞成像技术,通过测量不同细胞中标记位点的物理距离,统计细胞间的差异程度,以及母细胞与子细胞间的位点距离相关性;特异性方面,成像系统的测量误差仅为63nm,可精准区分细胞间的构象差异,细胞间位点距离差异可达2-4倍(文献未明确提供敏感性的ROC曲线数据)。
核心成果提炼显示,该染色质构象异质性状态可在单个细胞中维持1-10分钟,但不随细胞分裂遗传(Mann-Whitney p=0.181);其创新性在于首次通过直接的活细胞成像实验证明,染色质构象的细胞间异质性不具有跨细胞分裂的遗传能力,同时发现已知的染色质特征无法解释染色质压缩状态的差异,提示存在未知的调控因素。这一成果为细胞间转录异质性的起源提供了潜在机制,即染色质构象的动态异质性可能导致基因转录的细胞间差异,且这种差异不会传递给子代细胞,为表观遗传调控的研究提供了新方向。
