1. 领域背景与文献引入
文献英文标题:A plasmid-based system for expressing small interfering RNA libraries in mammalian cells;发表期刊:BMC Cell Biology;影响因子:未公开;研究领域:RNA干扰(RNAi)技术与哺乳动物细胞siRNA表达系统开发。
RNA干扰(RNAi)是1998年Fire等在秀丽隐杆线虫中首次发现的基因沉默机制,随后在植物中发现的转录后基因沉默(PTGS)也被证实是RNAi的同源过程。但哺乳动物体细胞中长双链RNA(dsRNA)会诱导干扰素反应,限制了RNAi的应用。2001年Elbashir等突破性发现,21-25nt的小干扰RNA(siRNA)可特异性沉默基因且不诱导干扰素反应,推动siRNA技术成为哺乳动物功能基因组学的核心工具。
现有siRNA表达系统中,基于pol III启动子的hairpin siRNA载体(如U6或H1启动子驱动)因成本低、可长期表达而广泛使用,但存在质粒不稳定(反向重复序列易导致细菌重组)、难测序、寡核苷酸成本高、siRNA含发夹环序列可能增加脱靶效应等局限性。此外,构建大规模siRNA文库用于功能筛选仍面临技术瓶颈。
本研究针对上述问题,开发了一种基于收敛性反向启动子的质粒系统(pHippy),旨在解决hairpin载体的缺陷,实现高效基因沉默并支持siRNA文库的快速构建,为功能基因组学研究提供更稳定、高效的工具。
2. 文献综述解析
作者系统梳理了RNAi技术的发展脉络,重点聚焦于哺乳动物siRNA应用的技术瓶颈:
1. RNAi基础研究:Fire等1998年线虫实验奠定RNAi的理论基础,Hamilton等1999年发现植物中21-22nt小RNA是PTGS的核心分子,Elbashir等2001年证实siRNA可避免哺乳动物干扰素反应,这些成果推动了siRNA技术的普及。
2. 现有载体系统的优缺点:hairpin siRNA载体成本低、可长期表达,但反向重复序列导致质粒不稳定、测序困难,且siRNA的发夹环序列可能引入脱靶效应;cDNA来源的siRNA文库虽已构建,但hairpin载体的技术难度限制了文库规模。
本研究的创新价值在于:
- 采用收敛性人H1和U6启动子(而非单个启动子驱动hairpin),避免反向重复序列,解决质粒不稳定问题;
- siRNA由两个启动子分别转录正义/反义链,无额外发夹环序列,减少脱靶效应;
- 载体设计BsmBl酶切位点,产生粘性末端,克隆效率近100%;
- 支持PCR快速构建表达盒,无需细菌转化,适合高通量制备;
- 可高效构建随机或cDNA来源的siRNA文库,用于功能筛选。
通过对比现有研究的局限性,作者明确pHippy系统的学术价值:为哺乳动物基因沉默提供更高效、稳定的工具,推动大规模功能基因组学研究。
3. 研究思路总结与详细解析
本研究核心目标是开发收敛性启动子的siRNA表达系统pHippy,验证其基因沉默效率,并建立文库构建方法。技术路线遵循“载体设计→功能验证(报告基因→内源基因)→高通量方法开发→文库筛选”的闭环逻辑。
3.1 pHippy载体的设计与构建
实验目的是设计可高效克隆siRNA序列的收敛性启动子载体。方法将人H1和U6启动子反向插入质粒,修饰启动子包含pol III终止信号(TTTTT,位于-5至-1位),并在-12至-6位引入BsmBl酶切位点(切割后产生3’端TTTT粘性末端,便于siRNA寡核苷酸的克隆)。结果显示,载体可高效克隆siRNA序列(超过25个双链寡核苷酸克隆效率近100%),载体结构如图1所示:
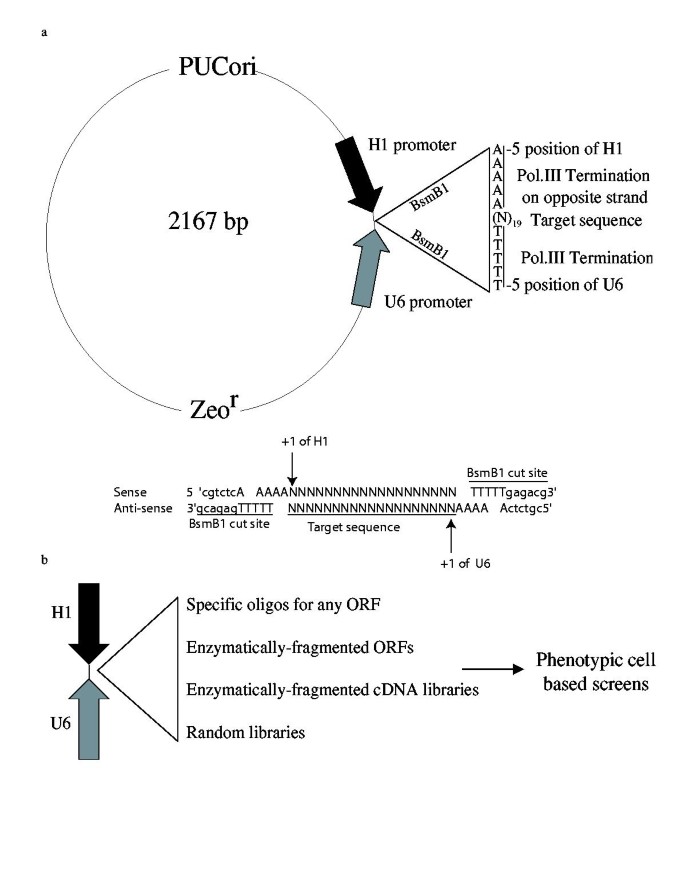
3.2 pHippy抑制报告基因表达的功能验证
实验目的是测试pHippy对报告基因的沉默效率及特异性。方法构建pHippyPGL3luc(针对PGL3 luciferase),与hairpin载体(U6PGL3lucHP、HIPGL3lucHP)比较,转染293T细胞后检测双荧光素酶活性;同时构建pHippyEGFP(针对EGFP)验证序列特异性。
结果显示:
- 空pHippy载体不影响PGL3表达(设为100%);
- pHippyPGL3luc的抑制效率显著高于hairpin载体(30ng DNA时,抑制效率是hairpin的2-4倍,n=3,P<0.05);
- pHippyEGFP仅抑制EGFP表达(共转染EGFP和DsRED时,EGFP荧光显著降低,DsRED无变化),验证序列特异性。
实验结果如图2所示:
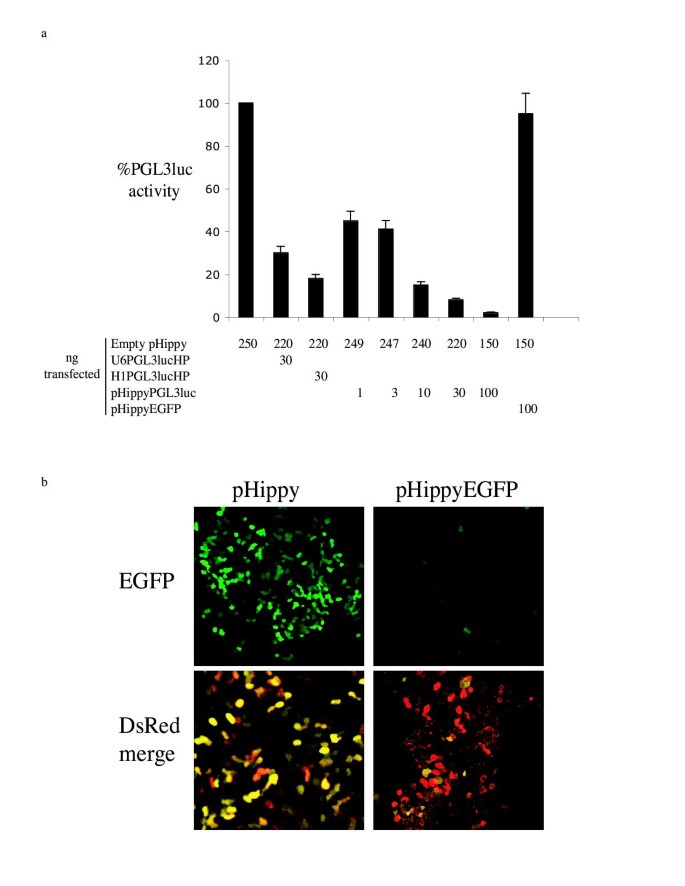
实验所用关键试剂:双荧光素酶检测试剂盒(Promega)、Lipofectamine 2000(Invitrogen)、pEGFPN1(Clontech)。
3.3 pHippy抑制内源基因表达的功能验证
实验目的是测试pHippy对哺乳动物内源基因的沉默效果。选择人LRP6基因(Wnt信号通路共受体,其功能可通过Wnt诱导的Super(8X)Topflash报告基因间接检测)作为靶标,构建5个pHippy-LRP6载体,通过以下步骤验证:
1. 筛选有效载体:转染293T细胞,抑制LRP6-Renilla融合蛋白(LRP6Rluc)的表达,结果5个载体中有3个抑制LRP6Rluc超过50%(n=3,P<0.05);
2. 验证内源功能:将有效载体转染293T细胞,Wnt3a条件培养基处理后,检测Super(8X)Topflash活性(反映Wnt通路活性)。结果显示,有效载体显著抑制Wnt3a诱导的报告基因活性(空载体组Wnt3a处理后活性升高100倍,有效载体组降至20-50倍)。
实验结果如图3所示:
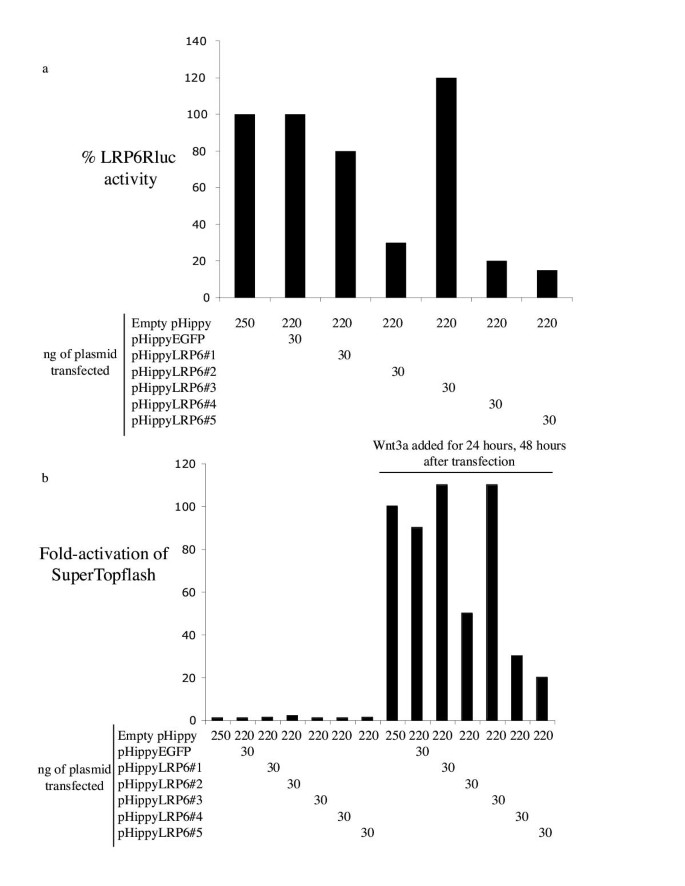
实验所用关键试剂:LRP6Rluc融合载体(自定义构建)、Super(8X)Topflash报告基因(Kaykas等2004年构建)。
3.4 高通量PCR法构建pHippy表达盒
实验目的是开发无需细菌转化的快速构建方法。设计三引物PCR体系:H1启动子引物(97nt)、基因特异性引物(包含U6/H1互补序列及靶基因序列)、U6反向引物(18nt),以U6启动子质粒为模板,通过touchdown PCR(30循环,退火温度60℃降至50℃)扩增表达盒。
结果显示,PCR可高效扩增正确大小的产物(针对PGL3的表达盒约200nt),转染293T细胞后剂量依赖地抑制PGL3表达(10ng PCR产物抑制30%,50ng抑制60%),虽效率略低于质粒,但证明高通量构建的可行性。
实验结果如图4所示:
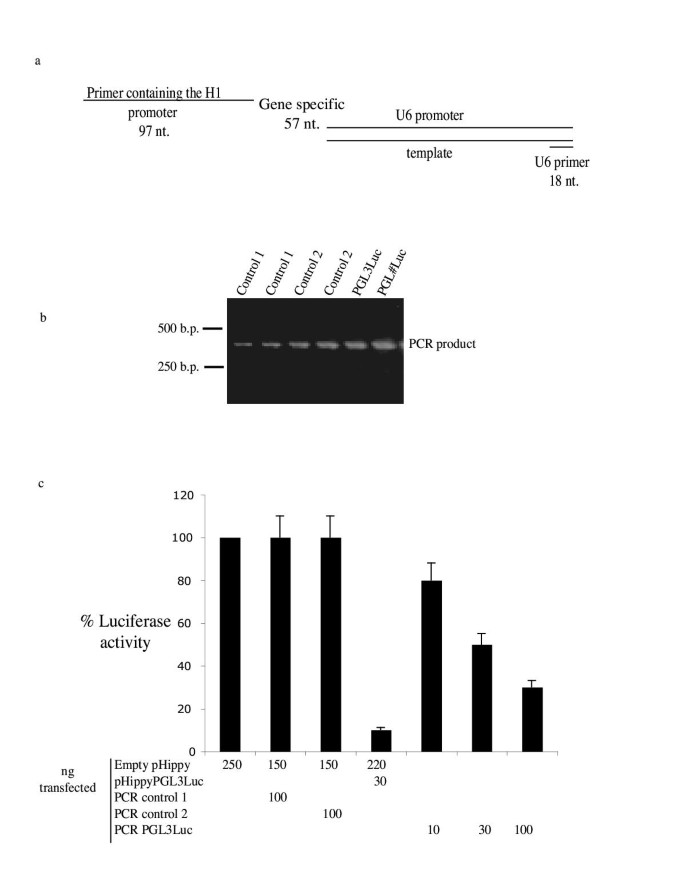
实验所用关键试剂:Advantage PCR试剂盒(Clontech)、NucleoSpin PCR纯化柱(Macherey-Nagel)。
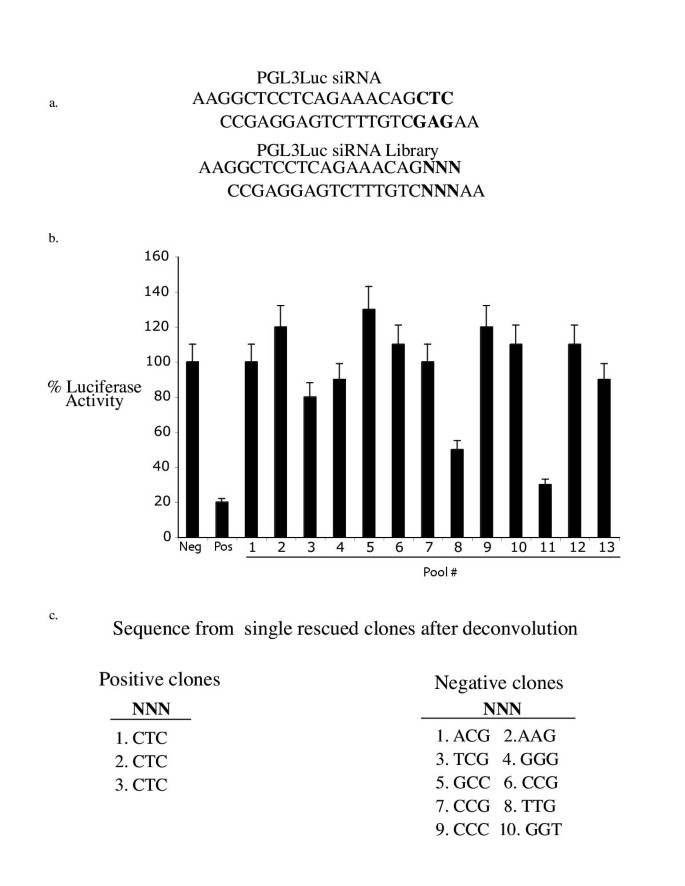
3.5 随机siRNA文库的构建与筛选
实验目的是验证pHippy构建文库并用于功能筛选的可行性。方法对PGL3的已知有效siRNA序列随机突变3个碱基(形成64种可能的文库),克隆到pHippy载体中;挑取130个细菌克隆,分成13个池(每池10个),筛选抑制PGL3的阳性池;对阳性池进行单克隆筛选并测序。
结果显示:
- 13个池中2个(池8、池11)显著抑制PGL3;
- 池8含1个有效克隆,池11含2个有效克隆;
- 测序证实有效克隆的siRNA序列与原序列一致,阴性克隆为随机突变。
实验结果如图5所示:
4. Biomarker研究及发现成果解析
本研究聚焦于siRNA表达系统的开发与功能验证,核心是构建高效的基因沉默工具,未涉及生物标志物(Biomarker)的筛选、验证或应用。因此,本部分无相关成果。
