1. 领域背景与文献引入
文献英文标题:Translocation of signalling proteins to the plasma membrane revealed by a new bioluminescent procedure;发表期刊:BMC Cell Biology;影响因子:未公开;研究领域:细胞信号转导(蛋白转位检测技术)。
细胞信号转导是调控细胞功能的核心过程,G蛋白偶联受体(GPCRs)和酪氨酸激酶受体(RTKs)被胞外配体激活后,会产生第二信使调控下游细胞功能,而可溶性效应蛋白向质膜的招募是信号调制、放大或抑制的关键步骤。过去数十年,针对第二信使生成的高通量检测方法已广泛应用于药物研发的化合物库筛选,但针对蛋白向质膜转位的高通量检测方法仍存在明显空白。现有检测方法中,传统的免疫细胞化学和亚细胞分馏结合免疫印迹法虽能证实转位过程的存在,但易因样本处理引入人工假象,且时空分辨率低、敏感性不足,仅适用于低通量的定性分析;绿色荧光蛋白(GFP)探针实现了活细胞内蛋白转位的实时监测,但荧光信号变化幅度有限,细胞内源性荧光会产生背景干扰,定量准确性不足;β-半乳糖苷酶互补法通过两个失活酶片段的互补恢复活性来检测转位,但需要同时构建靶蛋白与定位蛋白的嵌合报告基因,会对转位过程产生潜在影响,且仅适用于已知靶定位的蛋白研究,不适合大规模药物筛选。因此,开发一种简便、高灵敏度、适用于高通量的蛋白转位检测方法,对解析信号通路机制和靶向药物研发具有重要的学术价值与必要性。
2. 文献综述解析
本研究对领域内现有研究的评述按检测技术的发展脉络分类,涵盖传统生化检测方法、荧光探针技术及酶互补技术三类,系统梳理了各类方法的优势与局限性,明确了当前领域缺乏高效、通用的高通量蛋白转位检测技术的核心空白。
传统的免疫细胞化学和亚细胞分馏方法,能直观展示蛋白的亚细胞定位变化,但样本处理过程易引入人工假象,无法实现实时动态检测,时空分辨率较低,仅能完成低通量的定性分析,难以满足大规模研究或筛选需求。GFP荧光探针技术突破了传统方法的局限,实现了活细胞内蛋白转位的实时监测,但由于荧光信号变化幅度有限,且细胞内源性荧光会产生背景干扰,定量检测的准确性不足,限制了其在高通量筛选中的应用。β-半乳糖苷酶互补法通过酶片段互补恢复活性的原理检测转位,但需要同时构建靶蛋白与特定亚细胞定位蛋白的嵌合报告基因,会对转位过程产生潜在影响,且仅适用于已知靶定位的蛋白研究,无法覆盖未知靶定位的蛋白,不适合大规模药物筛选。
本研究创新性地利用水母发光蛋白(aequorin)的钙离子敏感性,结合质膜下区域与胞质的钙离子浓度差,构建了单报告基因的检测体系,无需复杂的双报告基因构建,且生物发光信号无内源性背景干扰,信号噪音比显著高于现有方法,填补了高通量蛋白转位检测技术的空白,为细胞信号转导研究和药物筛选提供了新的技术范式。
3. 研究思路总结与详细解析
本研究的核心目标是开发一种基于生物发光的蛋白向质膜转位检测方法,验证该方法的可靠性与高通量应用潜力,技术路线遵循“嵌合探针构建→转位功能验证→发光检测有效性验证→自动化高通量优化”的闭环逻辑,通过多环节实验逐步验证方法的可行性与性能。
3.1 嵌合探针构建
实验目的是构建融合水母发光蛋白的靶蛋白嵌合载体,为后续转位检测提供核心工具。方法细节为将β-抑制蛋白2(β-arrestin-2)和蛋白激酶CβII(PKCβII)的编码cDNA分别与水母发光蛋白(aequorin)的cDNA进行框内融合,构建得到βarr2-AEQ和PKCβ-AEQ两种嵌合报告基因载体,载体骨架采用pcDNA3真核表达载体。结果解读显示,成功构建了序列连贯的嵌合cDNA载体,经测序验证序列正确性,为后续功能验证实验奠定了基础。文献未提及具体实验产品,领域常规使用分子克隆相关试剂(如限制性内切酶、T4 DNA连接酶)及真核表达载体等。
3.2 嵌合探针转位功能验证
实验目的是确认水母发光蛋白标签不会影响靶蛋白的活性依赖型转位特性,保障检测方法的特异性。方法细节为采用HeLa细胞作为实验模型,该细胞内源性β-肾上腺素受体(βAR)表达量极低,将βarr2-AEQ与β2AR或空载体共转染,用40 μM异丙肾上腺素刺激1分钟;单独转染PKCβ-AEQ的细胞用1 μM佛波酯(PMA)刺激5分钟,通过免疫组化(IHC)检测嵌合蛋白的亚细胞定位变化,同时辅以亚细胞分馏结合免疫印迹验证(结果未展示)。结果解读显示,免疫组化结果(图2)显示,静息状态下βarr2-AEQ呈胞质弥漫分布,与β2AR共转染的细胞经异丙肾上腺素刺激后,约80%的细胞中βarr2-AEQ向质膜转位;PKCβ-AEQ在PMA刺激后,≥95%的细胞中嵌合蛋白从胞质转位至质膜,转位时间与已报道的两种蛋白的转位特性完全一致,说明水母发光蛋白标签未影响靶蛋白的转位功能。
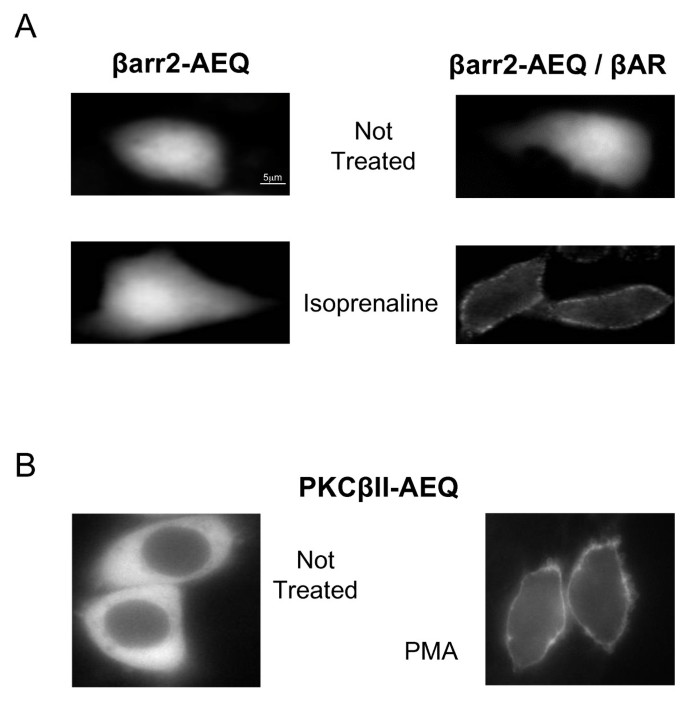
实验所用关键产品:兔抗arrestin多克隆抗体(Calbiochem,UK)、小鼠抗PKCβII单克隆抗体(Santa-Cruz Biotec.,CA,USA)、AlexaFluor 594标记的抗兔/抗鼠二抗(Invitrogen,Molecular Probes,CA,USA)、Zeiss LSM 510共聚焦激光扫描显微镜。
3.3 生物发光法检测蛋白转位有效性验证
实验目的是验证通过水母发光蛋白的发光变化可定量检测靶蛋白向质膜的转位,确认方法的定量准确性。方法细节为将HeLa细胞分别转染胞质定位对照探针(cyt-AEQ)、质膜内侧定位对照探针(SNAP-AEQ)、βarr2-AEQ+β2AR或PKCβ-AEQ,转染36小时后,在无钙缓冲液(KRB/EGTA)中孵育1小时并重构水母发光蛋白,随后用异丙肾上腺素刺激1分钟或PMA刺激5分钟,再恢复胞外钙离子浓度,用单孔发光仪检测发光信号。结果解读显示,质膜定位的SNAP-AEQ在钙恢复后的发光峰值比胞质定位的cyt-AEQ高10倍以上(图3);βarr2-AEQ在异丙肾上腺素刺激后的发光峰值为6951±797 cps(n=9),显著高于未刺激组的1730±356 cps(n=9);PKCβ-AEQ在PMA刺激后的发光峰值为44140±7858 cps(n=15),显著高于未刺激组的4115±1041 cps(n=12),刺激组的发光水平均接近SNAP-AEQ的水平,说明靶蛋白转位至质膜后,水母发光蛋白暴露于质膜下的高钙环境,发光信号显著增强,可用于定量检测转位过程。
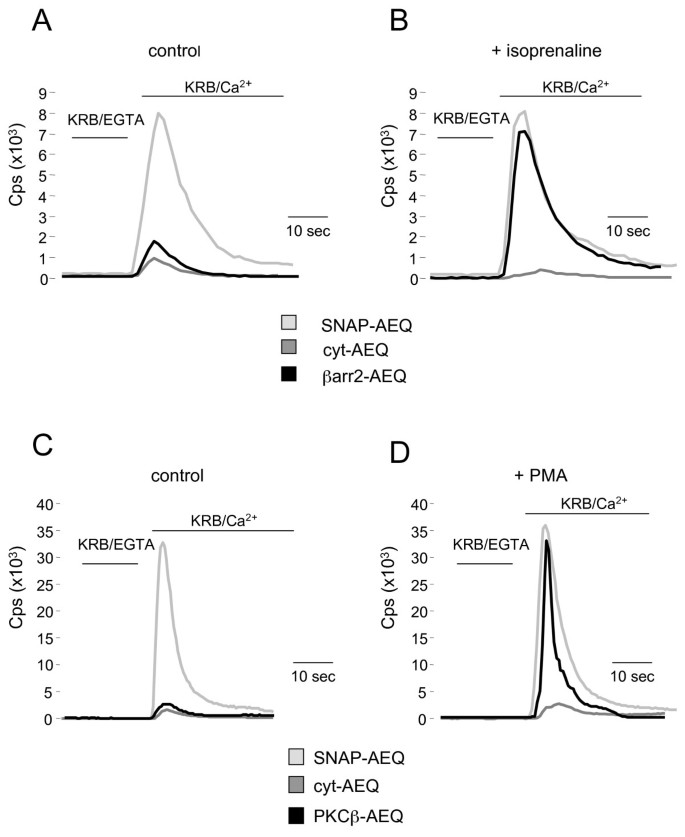
文献未提及具体实验产品,领域常规使用发光检测仪、细胞培养试剂及钙离子缓冲液等。
3.4 自动化高通量检测方法优化
实验目的是将该生物发光方法适配自动化高通量筛选平台,验证其动力学与剂量依赖性检测能力,拓展方法的应用场景。方法细节为采用24孔板培养HeLa细胞,转染βarr2-AEQ+β2AR,用机器人控制的发光读板仪进行自动化检测,分析异丙肾上腺素刺激后的发光峰值、转位动力学及剂量响应曲线。结果解读显示,自动化检测结果显示刺激组的发光峰值显著高于对照组(图4A、B);转位动力学分析显示,异丙肾上腺素刺激约30秒后达到最大响应,随后维持平台期(图4C);剂量响应曲线显示,异丙肾上腺素浓度低至10^-8 M时即可检测到转位信号,10^-4 M时达到最大响应,该曲线与之前用β-半乳糖苷酶互补法报道的βarr2与β2AR结合曲线高度一致,说明该方法可用于定量分析转位的动态过程与剂量依赖性,适用于高通量筛选。
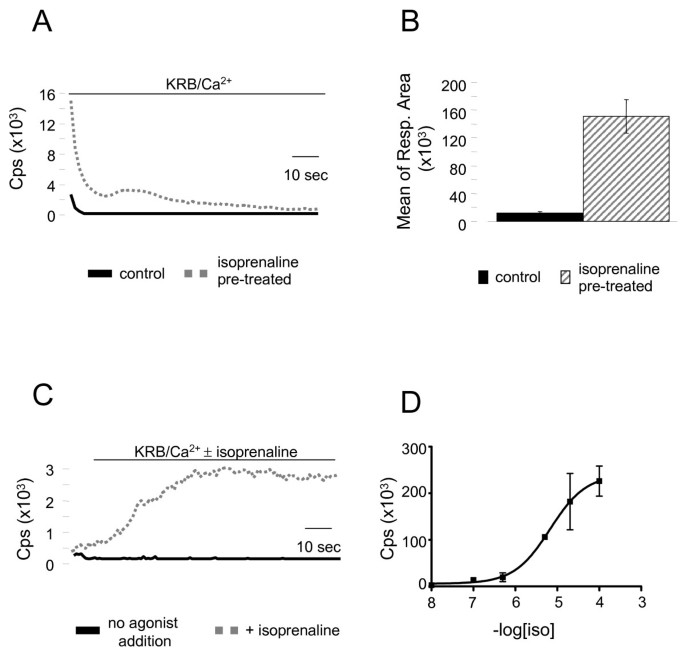
文献未提及具体实验产品,领域常规使用自动化发光读板仪、多孔细胞培养板及液体处理机器人等。
4. Biomarker研究及发现成果
本研究的核心“Biomarker”为基于水母发光蛋白的嵌合报告蛋白,作为蛋白向质膜转位的检测标志物,其筛选与验证逻辑遵循“载体构建→功能验证→发光信号验证→高通量适配”的完整链条,为细胞信号转导研究与药物筛选提供了新型检测工具。
Biomarker定位为重组的嵌合报告蛋白(βarr2-AEQ、PKCβ-AEQ),筛选逻辑是利用水母发光蛋白的钙离子敏感性,结合质膜下区域与胞质的钙离子浓度差(质膜下钙离子浓度比胞质高至少一个数量级),当靶蛋白转位至质膜时,嵌合蛋白暴露于高钙环境,发光信号显著增强,从而实现转位过程的定量检测。研究过程中,Biomarker的来源为分子克隆构建的真核表达载体,验证方法包括免疫组化验证转位功能、生物发光检测信号变化、自动化检测验证高通量性能;特异性表现为仅当靶蛋白转位至质膜时才会产生显著的发光信号,无内源性生物发光背景干扰,信号噪音比显著高于现有荧光检测方法;敏感性方面,可检测到低至10^-8 M的异丙肾上腺素刺激,样本量为n=9-15,文献未明确提供统计学显著性P值,但均值±标准误显示组间差异具有统计学意义。
核心成果为首次开发了基于单报告基因的生物发光蛋白转位检测方法,该方法不影响靶蛋白的转位特性,可实现实时动态检测与高通量筛选,适用于GPCRs、RTKs及其他非受体依赖的质膜招募蛋白的信号研究,为细胞信号转导机制解析与靶向药物筛选提供了高效、可靠的技术工具,具有重要的学术价值与应用潜力。该方法的自动化版本可提供转位过程的动力学与剂量依赖性信息,满足大规模药物筛选的需求,有望成为药物研发领域的核心技术之一。
