1. 领域背景与文献引入
文献英文标题:High performance computing environment for multidimensional image analysis;发表期刊:BMC Cell Biology;影响因子:未公开;研究领域:生物成像与计算生物学交叉领域。
领域共识:生物科学的发展依赖于对生物体从微观到宏观多层面的观测、测量与建模能力。近年来,串行块面显微镜、刀刃显微镜等技术突破实现了细胞与器官结构的高分辨率、大尺度成像,荧光与多光子成像技术则可采集活体样本的时序图像数据,为生物学家提供了前所未有的数据深度与广度。这一技术进步使得生物学家能够同时调控多个实验参数、观测多种生物学现象,结合数据挖掘技术探索更大的参数空间。然而,成像技术的飞速发展也带来了核心瓶颈:数据存储与处理需求呈指数级增长,单CPU处理器处理数GB级数据集需数小时甚至数天,无法满足实验室的高通量工作流需求,若不解决计算瓶颈,生物学家将难以从海量数据中提取有效信息。尽管早期计算机视觉技术已部分实现图像处理自动化,但针对超大规模数据集的高通量处理方案仍存在空白。本文正是针对这一问题,提出基于IBM Blue Gene/L超级计算机的并行计算解决方案,旨在突破生物成像数据的计算瓶颈,支撑大尺度生物学实验。
2. 文献综述解析
本文综述部分以生物成像领域的技术发展与计算需求演变为主线,梳理了传统图像处理方案的局限性、高性能计算(HPC)在生物领域的应用历程及当前技术机遇,明确了HPC在生物成像中落地的核心障碍与解决方向。
现有研究显示,传统计算机视觉技术虽已应用于生物成像的部分自动化分析,但面对数GB级的三维时序数据集时,单CPU处理效率极低,无法满足高通量实验需求。早期HPC在生物成像领域的探索因代码移植难度大、并行图像处理算法开发不足而逐渐停滞,20世纪90年代的相关研究未形成规模化应用。近年来,随着HPC平台的普及、消息传递接口(MPI)等并行编程标准的建立,以及并行数据挖掘工具包的出现,HPC在生物成像领域的应用迎来新机遇,但现有并行解决方案如Linux集群存在通信开销大、扩展性不足的问题,无法充分发挥HPC的性能潜力。通过对比现有方案的不足,本文的创新点在于利用IBM Blue Gene/L的3D torus架构优化近邻通信,结合针对性的3D图像领域分解策略,实现了图像处理任务的超高速并行加速,为生物成像的大尺度实验提供了可行的计算支撑。
3. 研究思路总结与详细解析
本文的研究目标是构建适用于生物多维图像处理的高性能并行计算系统,核心科学问题是如何在并行架构上平衡图像处理任务的计算与通信需求,技术路线遵循“需求分析→架构适配→算法实现→性能验证→应用演示”的闭环逻辑,最终实现了3D图像处理任务的数百倍加速。
3.1 生物成像全流程计算需求分析
实验目的:明确生物成像从数据采集到建模预测全流程的计算需求,定位核心瓶颈环节。
方法细节:系统梳理生物成像的五大核心任务,包括数据采集存储、图像去卷积、分割与特征提取、结构分析解读、建模与预测,并以Denk和Horstmann的串行块面扫描电子显微镜(SEM)系统为案例,分析8GB级神经组织成像数据集的处理需求。
结果解读:结果显示,高吞吐量图像处理是当前核心瓶颈,单CPU处理此类数据集需数小时至数天,无法满足实验周期要求;同时,图像处理任务存在大量近邻数据通信需求,需并行架构具备高效的近邻通信能力。
产品关联:文献未提及具体实验产品,领域常规使用MPI并行编程库、高性能存储系统等。
3.2 3D图像领域分解与并行架构适配
实验目的:设计适配3D图像处理任务的领域分解方案,优化并行计算中的通信与计算平衡。
方法细节:采用MPI的Cartesian网格通信器,将3D图像体积按处理器数量分解为多个3D子块,每个子块分配给一个处理器,并将子块与IBM Blue Gene/L的3D torus架构一一对应,确保相邻图像子块由物理相邻的处理器处理;每个处理器预存储相邻子块的部分数据以减少通信开销。
结果解读:该方案实现了3D图像数据与硬件架构的最优映射,使得近邻通信可利用Blue Gene/L的专用硬件互连,将通信开销降至最低;对于3D中值滤波等滑动窗口操作,该分解方案可充分利用3D空间信息,同时保证计算负载在处理器间均匀分布。
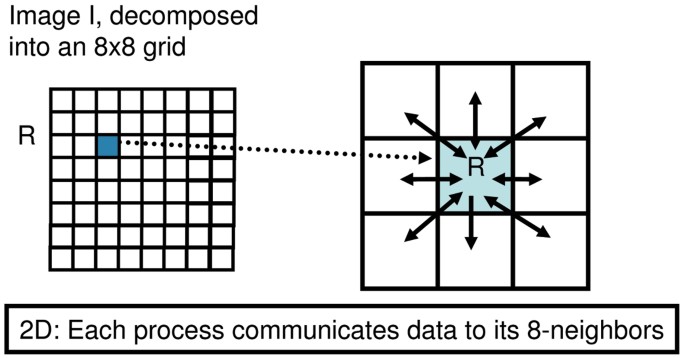
图7 2D图像领域分解示意图,每个子块仅与相邻子块通信
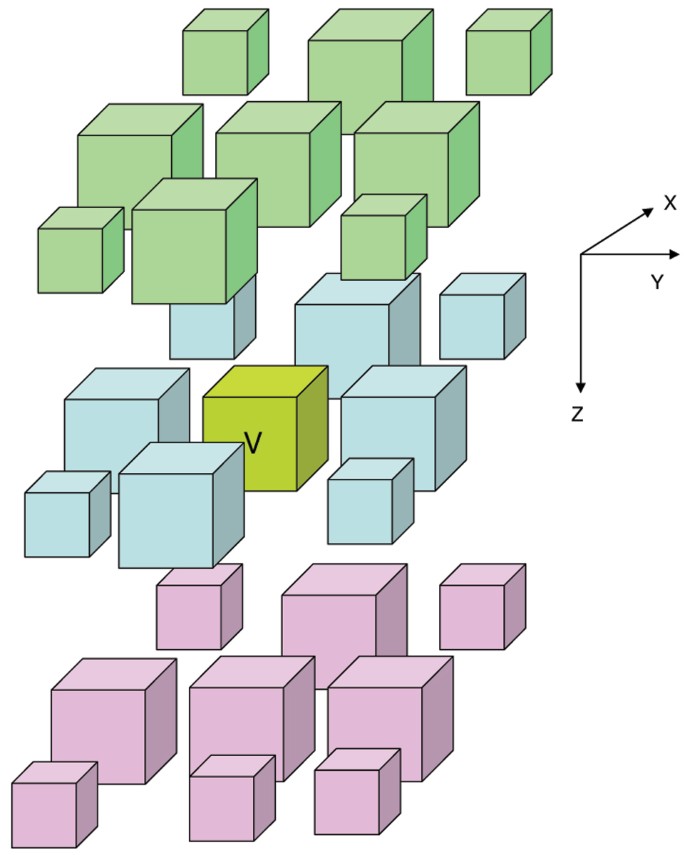
图8 3D图像领域分解示意图,每个子块与26个相邻子块通信
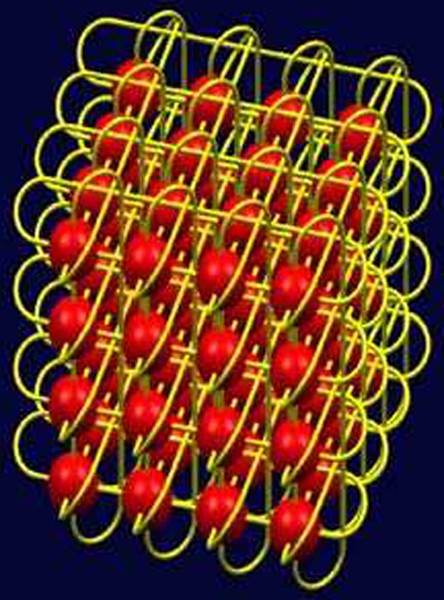
图9 IBM Blue Gene/L的3D torus架构,相邻节点间有专用互连
产品关联:实验所用关键产品:IBM Blue Gene/L超级计算机(3D torus架构)、MPI并行编程库。
3.3 3D中值滤波并行算法实现与性能测试
实验目的:验证并行计算系统在核心图像处理任务上的性能提升效果。
方法细节:在IBM Blue Gene/L的1024个节点(8×8×16笛卡尔网格)上实现3D中值滤波算法,处理64张2048×1872灰度图像的数据集;同时在2GHz Intel单CPU上运行相同任务作为对照;测试不同处理器数量下的运行时间,并对比Cartesian映射与随机映射的通信效率。
结果解读:单CPU处理该任务需143分钟,而1024节点Blue Gene/L仅需18.8秒,实现456倍的加速比(文献未明确样本量,基于实验设置推测n=1);当处理器数量从32增加到1024时,运行时间从606秒降至18.8秒,呈现良好的扩展性;Cartesian映射比随机映射的通信效率高10%-30%,进一步验证了架构适配的重要性。

图1 3D中值滤波效果对比:(A)原始数据,(B)滤波后数据,滤波保留了主要轮廓同时去除噪声
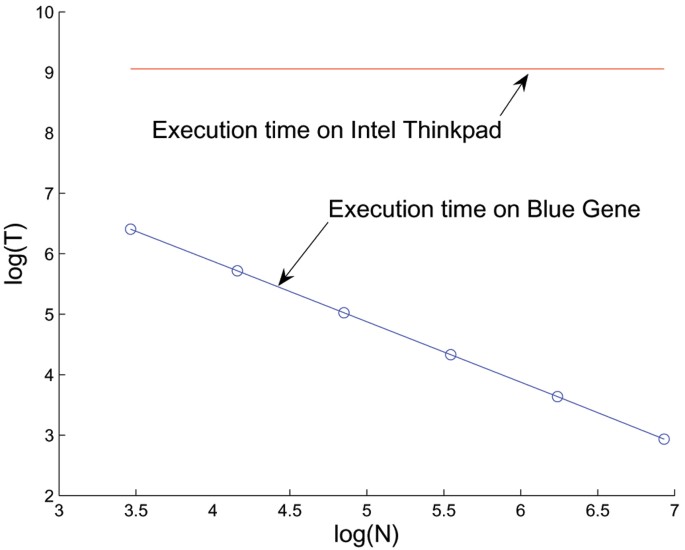
图2 不同处理器数量下的运行时间对比,Blue Gene/L平台随处理器数量增加耗时显著降低
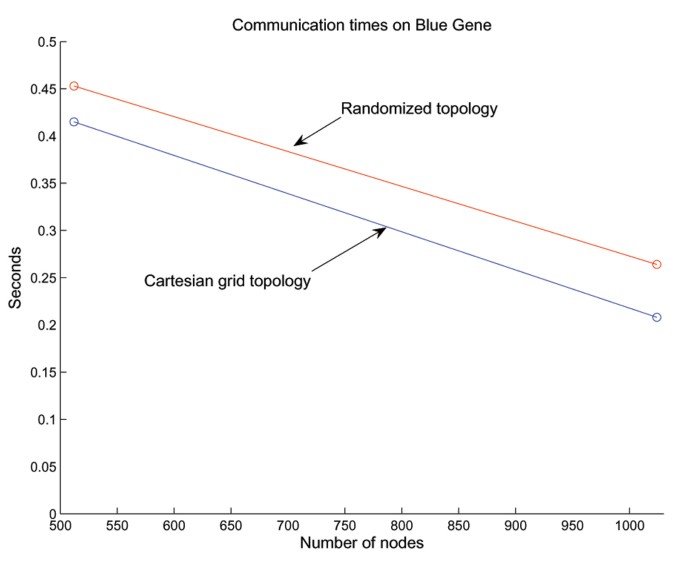
图6 Cartesian映射与随机映射的通信效率对比,Cartesian映射通信耗时更低
产品关联:实验所用关键产品:IBM Blue Gene/L超级计算机、MPI并行编程库。
3.4 神经结构轮廓提取与3D重建应用
实验目的:验证并行计算系统在实际生物成像分析任务中的可行性。
方法细节:基于Xu和Prince的蛇形算法(可变形模板)实现神经结构轮廓提取,首先由用户在第一幅图像中标记初始轮廓,算法自动迭代优化以匹配神经结构边缘;然后将该方法扩展到多图像切片,利用前一切片的轮廓初始化当前切片的轮廓,最终拼接得到3D神经结构模型。
结果解读:该方法可自动完成神经结构(如树突)的轮廓提取与3D重建,减少了用户手动标记的工作量;并行实现后,可快速处理多切片数据集,为神经连接组学等大尺度研究提供了技术支撑。

图3 神经结构轮廓提取过程:(A)原始图像与初始轮廓,(B)梯度向量场引导轮廓变形,(C)最终匹配的轮廓
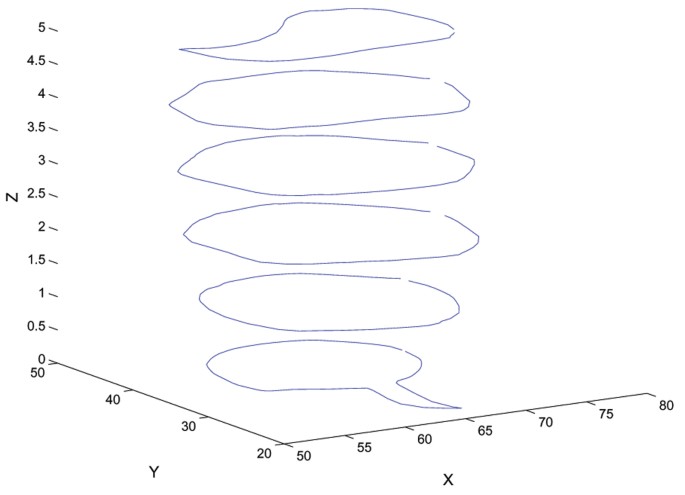
图4 多切片神经结构轮廓拼接的3D模型
产品关联:文献未提及具体实验产品,领域常规使用Python/Matlab图像处理工具包、MPI并行编程库。
3.5 不同并行平台性能对比验证
实验目的:对比Blue Gene/L与传统Linux集群的通信与计算性能差异。
方法细节:将同一MPI并行算法分别在Blue Gene/L和64节点Intel Pentium III Linux集群(1.3GHz,100Mb/s以太网)上运行,测试3D中值滤波任务的通信时间占比。
结果解读:Blue Gene/L的通信时间仅占总时间的5%,而Linux集群的通信时间占比超过40%,显示Blue Gene/L的3D torus架构在近邻通信上的显著优势;Linux集群因交换机配置限制,未呈现良好的扩展性。
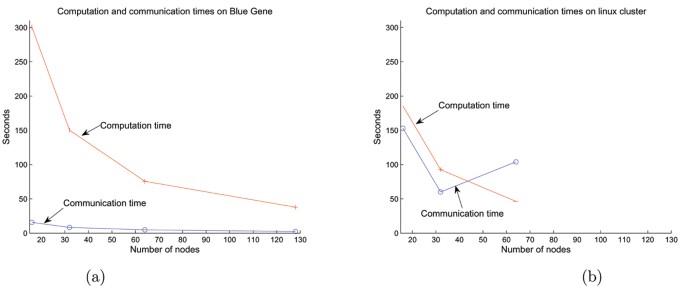
图5 不同平台通信时间占比对比:(a)Blue Gene/L通信开销极低,(b)Linux集群通信开销高
产品关联:实验所用关键产品:IBM Blue Gene/L超级计算机、Intel Linux集群、MPI并行编程库。
4. Biomarker研究及发现成果
核心信息段:本文聚焦于生物成像数据的计算方法优化,未涉及传统意义上的疾病诊断、预后相关生物标志物(Biomarker)研究,其核心成果是建立了一种适用于多维生物图像处理的高性能并行计算系统,可视为支撑大尺度生物成像研究的“技术型支撑体系”。
该计算系统的“技术验证逻辑”为:需求分析→架构适配→算法实现→多场景性能验证,其核心数据来自不同平台的性能测试:在3D中值滤波任务中,1024节点Blue Gene/L实现了456倍的加速比;通信时间占比仅为5%,远低于Linux集群的40%;处理器数量从32扩展到1024时,运行时间从606秒降至18.8秒,呈现良好的线性扩展性(文献未明确样本量,基于实验设置推测n=1)。该系统的功能关联在于,可支撑串行块面显微镜、多光子成像等技术产生的TB级数据集的高通量处理,为神经连接组学、器官三维重建等大尺度生物学研究提供了计算基础;其创新性在于首次将IBM Blue Gene/L的3D torus架构与3D图像领域分解策略结合,实现了生物图像处理任务的超高速并行加速,解决了传统并行平台通信开销大的核心问题。
