1. 领域背景与文献引入
文献英文标题:Transcriptome and DNA methylation profiling during the NSN to SN transition in mouse oocytes;发表期刊:BMC Molecular and Cell Biology;影响因子:未公开;研究领域:卵母细胞发育与表观遗传学。
哺乳动物卵母细胞发育后期会经历非包围核仁(NSN)向包围核仁(SN)的染色质重构,伴随转录沉默,这是卵母细胞获得发育能力的关键步骤。领域发展关键节点包括:1999年发现NSN-SN转变与转录沉默相关;2016年scM&T-seq技术实现单细胞核转录组与DNA甲基化并行分析;2020年报道aged卵母细胞SN比例升高。当前研究热点聚焦于卵母细胞成熟的转录后调控与表观遗传修饰(如DNA甲基化、组蛋白修饰),未解决的核心问题是NSN-SN转变伴随的转录组与DNA甲基化动态变化及其功能关联——现有研究多关注单一分子层面,缺乏两者的整合分析,且对SN卵母细胞中DNA甲基化的调控机制不清。
针对这一空白,本研究旨在解析NSN-SN转变中的转录组与DNA甲基化变化,揭示两者的协同调控关系,为理解卵母细胞发育能力的分子基础提供新依据。
2. 文献综述解析
文献综述的核心评述逻辑:作者按卵母细胞发育阶段(NSN→SN)和分子层面(转录组、DNA甲基化)分类,系统梳理现有研究的进展与局限。
现有研究的关键结论:① NSN-SN转变与转录沉默高度相关,但两者可通过基因敲除(如Zfp36l2 KO)解偶联;② scRNA-seq比bulk RNA-seq更能检测NSN-SN的转录组差异( reproducibility更高);③ 卵母细胞生长过程中DNA甲基化重新建立(依赖DNMT3A/3L与H3K36me3),但NSN-SN转变中的甲基化变化未详细报道。技术方法优势:scM&T-seq可同时分析单细胞核的转录组与DNA甲基化,解决了卵母细胞样本量少的问题;局限性:现有研究未整合转录组与DNA甲基化数据,且对NSN-SN转变中DNA甲基化与组蛋白修饰的关联不清楚。
本研究的创新价值:① 首次用scM&T-seq技术整合分析NSN-SN转变的转录组与DNA甲基化动态;② 开发基于100个核心基因的转录组classifier,实现对卵母细胞染色质状态的无创推断;③ 发现SN卵母细胞中H3K36me3+H3K27me3(通常互斥)的独特组蛋白修饰组合,关联DNA甲基化的晚期建立,为表观遗传可塑性提供新线索。
3. 研究思路总结与详细解析
3.1 整体框架
研究目标:解析小鼠卵母细胞NSN-SN转变的转录组与DNA甲基化动态;核心科学问题:NSN-SN转变中转录组与DNA甲基化的变化规律及功能关联;技术路线:卵母细胞分类→scM&T-seq建库→转录组分析(差异基因+classifier)→DNA甲基化分析(DMRs)→组蛋白修饰关联验证,形成“表型-分子-功能”的闭环。
3.2 卵母细胞收集与分类
实验目的:获得NSN与SN阶段的生发泡(GV)卵母细胞;方法细节:从2只12周龄C57Bl6/Babr小鼠卵巢分离37个GV卵母细胞,去除卵丘细胞后用5μM Hoechst(Abcam,33343)染色10min,共聚焦显微镜分类(NSN:无核仁环;SN:完整核仁环);结果解读:筛选得到13个NSN、2个中间型、22个SN卵母细胞,排除2个 outliers后,保留9个NSN和16个SN用于后续分析;实验所用关键产品:Hoechst抗体(Abcam,33343)、M2培养基(Sigma-Aldrich,M7167)。
3.3 scM&T-seq文库构建与测序
实验目的:并行获得单细胞核的转录组与DNA甲基化数据;方法细节:用scM&T-seq分离mRNA(poly-dT磁珠)与DNA,mRNA反转录为cDNA(SuperScript II)后用Nextera XT Kit构建scRNA-seq文库;DNA经亚硫酸氢盐处理后用Bismark流程构建scBS-seq文库,Illumina NextSeq500测序(RNA-seq:150bp单端,3.5M reads/样本;BS-seq:75bp双端,19M reads/样本);结果解读:获得9个NSN和16个SN的RNA-seq数据,及12个NSN和28个SN的BS-seq数据(覆盖~10%-22%基因组CpG位点);实验所用关键产品:Nextera XT Kit(Illumina)、Bismark软件。
3.4 转录组差异分析与classifier开发
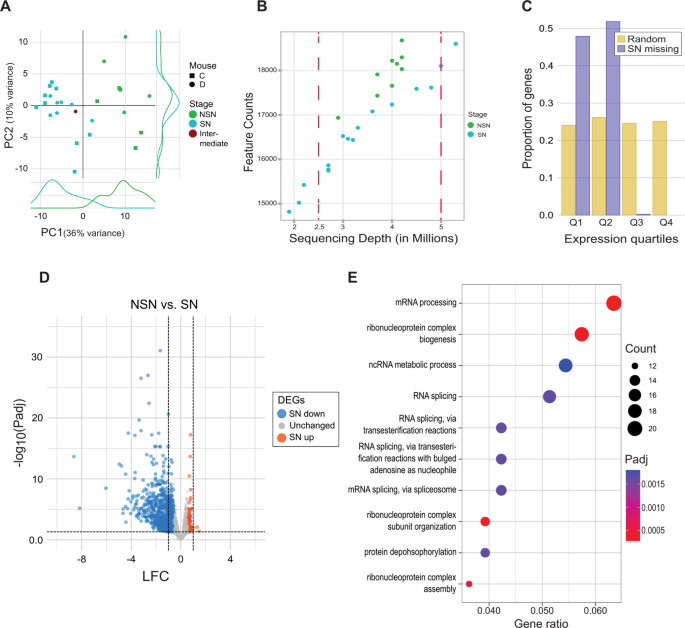
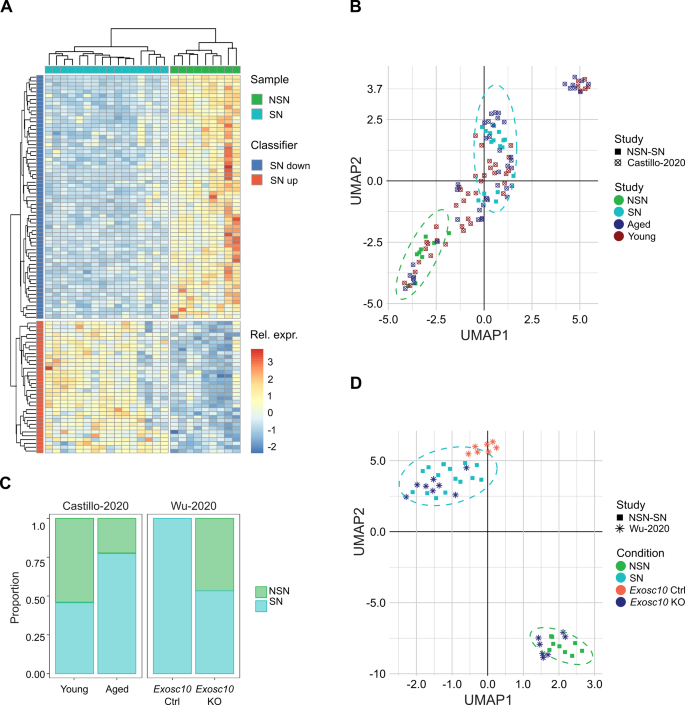
实验目的:分析NSN-SN的转录组差异并开发染色质状态分类器;方法细节:用DESeq2分析差异表达基因(DEGs,padj<0.05,LFC>|1|),从576个下调基因中筛选100个核心DEGs(结合fold-change与FDR)构建classifier,用PCA、UMAP验证其有效性;结果解读:① NSN卵母细胞比SN多检测到1164个转录本(n=9/16,P=0.0012);② 576个基因下调(富集mRNA加工、核糖核蛋白组装等过程);③ classifier可有效区分NSN/SN:如aged(>40周)卵母细胞SN比例(72%)显著高于年轻(12周,45%)(n=87,P<0.05),Exosc10 KO卵母细胞SN比例(30%)显著低于野生型(85%)(n=42,P<0.01);实验所用关键产品:DESeq2软件、UMAP软件。
3.5 DNA甲基化动态与DMR鉴定
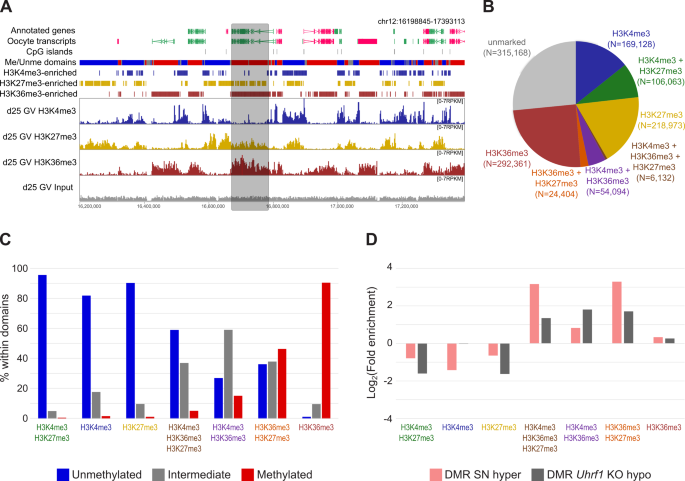
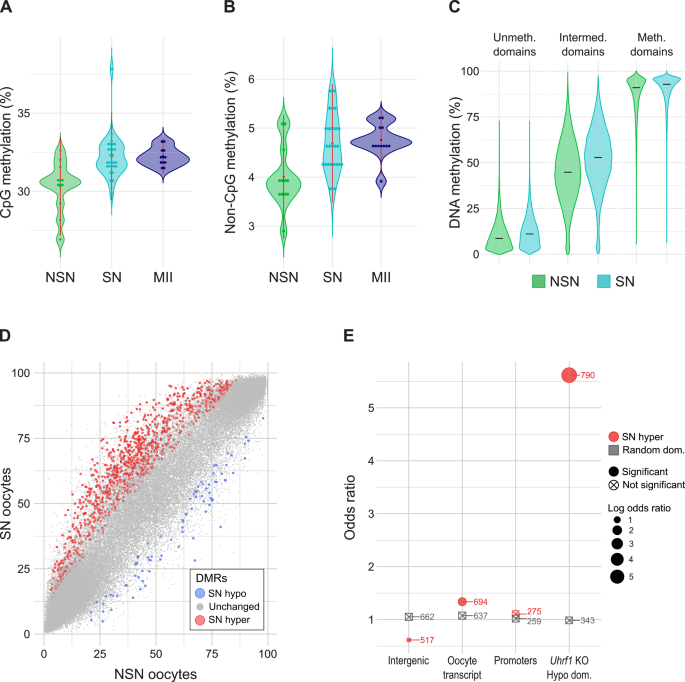
实验目的:解析NSN-SN的DNA甲基化变化;方法细节:用Bismark定量CpG甲基化水平,通过伪bulk分析(3组4个卵母细胞,100次迭代)鉴定差异甲基化区域(DMRs,甲基化差异>10%且重复50%以上);结果解读:① SN卵母细胞全局甲基化(32.3%)高于NSN(30.3%)(n=12/28,P<0.05),且与MII卵母细胞(成熟卵)水平一致;② 中间甲基化区域(25%-70%)变化最显著(占DMRs的68%);③ 1064个DMRs在SN中高甲基化(富集Uhrf1 KO卵母细胞的低甲基化区域,odds ratio=5.62,P<0.001);实验所用关键产品:Bismark软件。
3.6 组蛋白修饰关联分析
实验目的:探究DNA甲基化变化与组蛋白修饰的关系;方法细节:整合本研究的H3K36me3 ChIP-seq数据与公共H3K4me3、H3K27me3数据,分析DMRs的组蛋白修饰富集;结果解读:SN高甲基化DMRs显著富集H3K36me3+H3K27me3组合(通常互斥),这类区域多为中间甲基化(占72%),提示其染色质可塑性高(可能处于H3K27me3向H3K36me3的转变中);实验所用关键产品:H3K36me3抗体(Diagenode,C15410192)、ChIP-seq文库构建试剂盒(未提及具体品牌,领域常规使用抗组蛋白修饰抗体)。
4. Biomarker研究及发现成果解析
Biomarker定位与筛选逻辑
本研究的Biomarker包括两类:① 转录组classifier的100个核心基因(如Mrpl4、Rps27a等mRNA加工相关基因),筛选逻辑为“NSN-SN差异表达→文献重叠验证→classifier性能测试”;② SN高甲基化DMRs(如chr1:1000000-1002000区域),筛选逻辑为“伪bulk分析→Uhrf1 KO重叠验证→组蛋白修饰富集”。
研究过程详述
- 转录组classifier:来源为NSN-SN的差异表达基因(DESeq2筛选),验证方法为UMAP聚类(测试aged、Exosc10 KO等模型),特异性:能准确区分不同模型的NSN/SN比例(如Sall4 KO卵母细胞SN比例<10%,野生型>80%);敏感性:对scRNA-seq(准确率85%)与bulk RNA-seq(准确率70%)均有效。
- SN高甲基化DMRs:来源为scBS-seq数据,验证方法为与Uhrf1 KO低甲基化区域重叠(重叠率45%,odds ratio=5.62,P<0.001),特异性:富集卵母细胞转录本区域(占62%),而非启动子或基因间区;敏感性:检测到1064个DMRs(占总区域的0.9%)。
核心成果提炼
- 转录组classifier:可作为卵母细胞染色质状态的无创 Biomarker,用于评估基因敲除或aged模型的NSN-SN比例(如Exosc10 KO卵母细胞SN比例降低,提示发育能力受损);
- SN高甲基化DMRs:作为表观遗传 Biomarker,关联H3K36me3+H3K27me3组合,提示这些区域是染色质可塑性热点(可能参与卵母细胞向胚胎的转变);
- 创新性:首次开发卵母细胞染色质状态的转录组classifier,发现SN卵母细胞中独特的组蛋白修饰关联DMRs;
- 统计学结果:classifier验证中aged卵母细胞SN比例显著高于年轻(χ²=12.3,P<0.001),DMRs与Uhrf1 KO重叠的odds ratio=5.62(95% CI 4.8-6.5,P<0.001)。
图片插入(对应实验环节)
3.4 转录组分析结果
3.4 classifier验证结果
3.5 DNA甲基化分析结果
3.6 组蛋白修饰关联结果